Még valamikor a 19. sz. végén, a kor egyik legjelesebb brit zoológusa, D’Arcy Thompson azzal szórakoztatta karácsonykor gyerekeit, hogy egy gumilapra kutyafigurát rajzolt és azt különböző irányba húzogatva egyik kutyafajtából csinált egy másikat. Thompson kvintesszenciális viktoriánus tudós volt és zoológia mellett nagyon érdekelte a matematika és fizika is (meg történetesen a klasszikus görög irodalom) és a gumihúzogatás a gyerekek szórakoztatásán túl egy mélyebb tudományos gondolat fizikai megtestesülése is volt. Utóbbi szerint az egyes élőlények kinézetét (a biológiai anyag formáját) elsősorban fizikai erők alakítják.
Ezt az elméletet öntötte aztán 57 évesen könyvformába “On Growth and Form” című művében, ami 1917-ben jelent meg és sokan máig a 20. század egyik legfontosabb biológiai témájú művének tartják. Pedig D’Arcy elsődleges szándéka, miszerint alternatívát kínáljon az öröklődés-alapú darwini evolúciós elméletnek, sosem kapott kellő támogatást. És mégis kellő mennyiségű érdekes gondolat került bele a műbe, hogy számos egymást követő kutatói generációt volt képes inspirálni. (Akit érdekelnek Thompson életének egyéb részletei, annak mindenképpen ajánlom Stephen Wolfram igazi fanboy-lelkesedéssel megírt hosszú írását.)

A karácsonyi gumikutyák logikája az “On Growth and Form” utolsó előtti fejezetében köszön aztán vissza, de itt (elsősorban) nem kutyákat, hanem halakat találunk. És ezek, a fent is látható rajzok lettek később a legikonikusabbak D’Arcy Thompson főművéből.
Mert bár az öröklődés fogja végső soron a testek alakját formálni, de azt nagyon jól ismerte fel Thompson, hogy bizonyos testrészek evolúciós átalakulása felfogható mint egy koordinátarendszer-torzuláshoz vezető növekedésbeli változás. Ezeknek pedig mind biológiai (genetikai) oka van, legyen az mondjuk a tacskók rövid lába, vagy a zsiráfok hosszú nyaka.
A halak esetében tényleg rengeteg ilyen típusú “transzformációt” figyelhetünk meg (persze nem teljesen véletlenül, hiszen a gerinces fajok fele az hal), és talán az egyik legérdekesebb közülük a repülőhalak mellúszójának esete.
Amiben egy tipikus repülőhal igazán különleges, az mellúszójának a mérete, amely egyben azt a relatíve nagy felületet is biztosítani tudja, ami ezeknek a fajoknak a siklórepüléséhez szükséges (és egy ilyen siklás akár 400 m hosszan is tarthat, miközben a magasságukat és a siklás irányát is szabályozzák). Hogy mi lehet ennek a megnagyobbodott mellúszónak az eredete, annak eredt a nyomába a Harvardon dolgozó Matthew Harris csoportja.
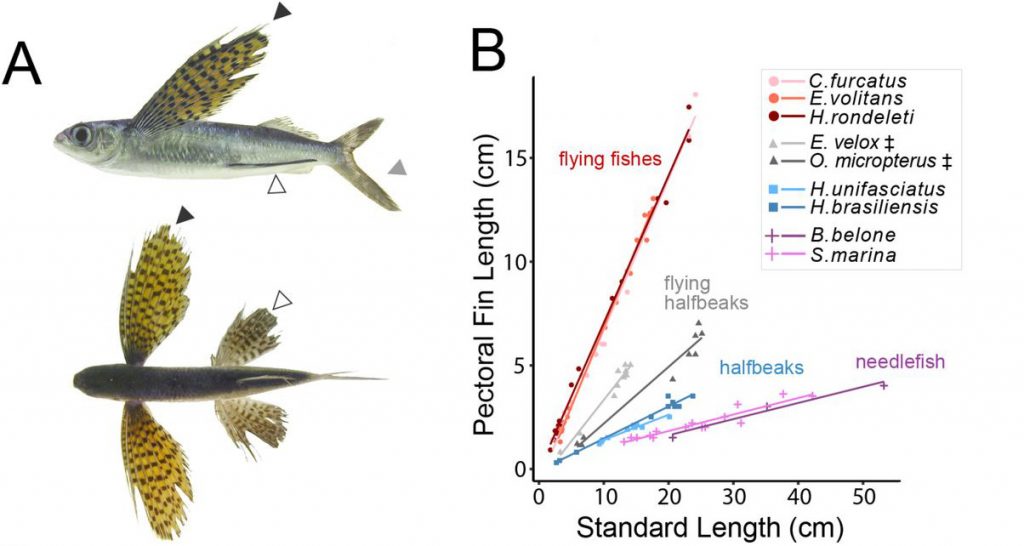
Az egyelőre csak preprint formájában elérhető kutatásban Harris-ék a repülőhalakat is magukba foglaló Beloniformes (magyarul Makrahalalakúak) rend 35 különböző fajának a fehérjekódoló, illetve konzervált, de nem kódoló szekvenciáját határozták meg. (Durva közelítéssel ezek a lényegi részei a genomnak, persze azért ez itt egy szükséges egyszerűsítés, ami ahhoz volt szükséges, hogy ennyi fajra költséghatékonyan meg lehessen oldani a szekvenálást.) Ezek segítségével meg lehetett becsülni, hogy milyen folyamatokhoz kapcsolódóak azok a fehérjekódoló gének, amelyek (feltehetőleg az adaptáció miatt) gyorsabban evolválódtak a repülőhalakban.
A gyorsan változó gének közt vannak olyanok, amelyek mondjuk az egyensúlyért felelős vesztibuláris rendszerhez kapcsolhatók, egyes izomfejlődésben fontos gének és számos olyan gén is, amelyet ilyen-olyan módon, más fajokban korábban már az uszony/végtag fejlődéséhez kapcsoltak. Ezek azok a gének, amelyeket jelen tudásunk alapján várnánk is, hogy változzanak, de persze egy ilyen összevetésben kijönnek olyan találatok is, amelyek általánosabb funkciót betöltő fehérjék és amelyek kevésbé “gyanúsak” egy ilyen esetben. Itt vannak például kálium-csatornákat (kcnk5a, kcnk9), vagy aminosav-transzportban fontos fehérjéket kódoló gének (lat4a, lat4b), amelyeket egy tipikus genomikai elemzésben még 4-5 évvel ezelőtt is a “futottak még” kategóriába soroltak. És ez az a pont ahol Harrisék csoportja szerencsére a genomikát egy jó adag (zebrahal)genetikával keverte össze, amitől igazán érdekes lett a történet.

Harris korábban a Nobel-díjas Christiane Nüsslein-Volhard egyik nagyon sikeres poszt-dokja volt és maga is részt vett abban a genetikai screen-ben, ahol olyan mutációkat kerestek, amelyek a felnőtt állatoknak változtatják meg valamiképpen a külalakját – mintázatát, pikkelyzetét, vázrendszerét, vagy épp az úszók méretét. Ez tipikusan olyan projekt volt, amihez egyrészt szükséges volt az a támogatás, amit egy Nobel díjas biztosítani tud, másrészt logikusan következett Nüsslein-Volhard (vagy, ahogy mindenki ismeri, Yani) korábbi munkáiból. Nobel díját Yani még azért a fantasztikus munkákért kapta, amit Eric Wieschaus-al együtt a korai muslica embriók fejlődésében szerepet játszó gének feltérképezése során végeztek (és ami az alapja minden fejlődésgenetika kurzusnak), majd ezután lényegében ugyanezt a munkát kiterjesztette zebrahal embriókra is. Ez volt ebben a fajban az egyik első komolyabb genetikai screen és egyben az egyik kulcsmomentum abból a szempontból is, hogy a George Streisinger által megálmodott modellszervezet valóban be tudott futni. De míg az embrionális genetikai screenek (főleg, ha muslicáról van szó), viszonylag gyorsak, addig ebben a legutóbbi kísérletsorozatban hónapokat, ha nem éveket kellett várni, amíg egyáltalán kiderült, hogy valóban új fenotípussal álunk szemben. Ebben a munkában játszott aztán fontos szerepet Harris.
Persze ekkor nem csak új mutánsokat kerestek, hanem a korábbi screenekből származó mutációkat is igyekeztek feltérképezni, így például azt is megérteni, hogy a nagyon eredetien longfin (lof) és another longfin (alf) névre keresztelt mutánsok, amelyek a nevüknek megfelelően meghosszabbodott úszókkal dicsekedhettek, milyen mutációknak köszönhetik létüket.
Az eredmény meglepő volt, hiszen ahogy először az alf esetében, majd később a lof-nál is kiderült, hogy a nagyobb úszóméretért kálium csatornák felelősek (előbbinél kcnk5b, utóbbinál pedig kcnh2a érintett). Mivel ezeket a csatornákat leginkább az idegrendszerben betöltött szerepük miatt tanulmányozták korábban, meglehetősen váratlan volt hogy megnövekedett expressziójuk eredményeképpen megnövekedett úszókat kaphatunk és egyben rávilágított, hogy a bioelektromos jelátvitel még sok más szövetben játszhat fontos szerepet.
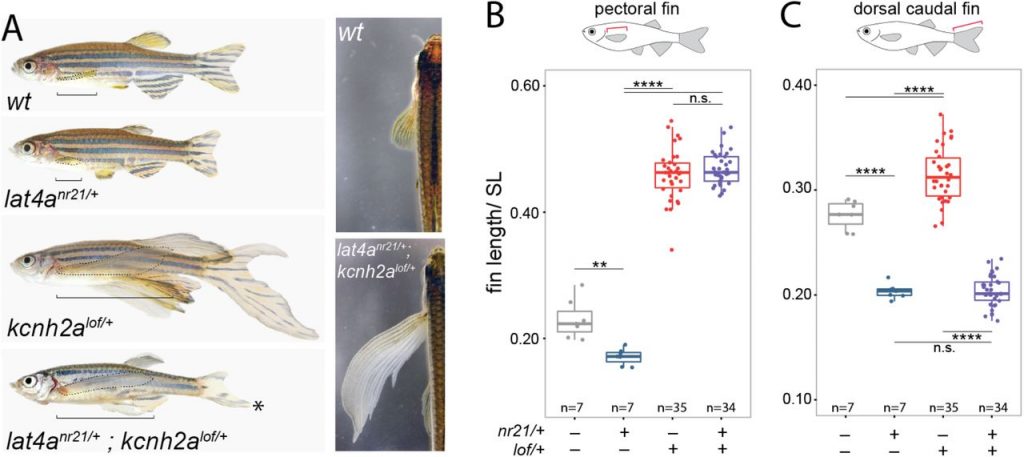
Harris csoportja is a kcnh2alof mutációból indult ki (ők maguk is feltérképezték a mutáció helyét), hiszen ezek az úszók hasonlítanak leginkább a repülőhalak megnövekedett úszóira. Ugyanakkor a lof halakban a farokúszó mindkét része is megnyúlt ez pedig a repülőhalaknál nincs így. Ezért egy ellentétes hatású mutációt kerestek, egy olyant, aminek következtében rövidebbek lesznek az úszók és végül a nem túl fantáziadús nr21 “személyében” akadtak egy ilyen fenotípust okozó, domináns mutációra.
Az nr21 esetében a mutáció a lat4a génbe esett (itt is egy expressziót növelő, funkció-nyeréses mutációról van szó) és itt már érthető is, hogy miért ér össze a repülőhalak és a fura uszonyú zebrahal-mutánsok története, hiszen, ahogy fentebb írtam pont ilyen típusú gének mentek keresztól felgyorsult evolúción a repülőhalak genomjában.
Persze, ha van egy mutációnk, ami hosszabb úszókat hoz létre és egy másik, ami meg rövidebbet, adja magát a kérdés, hogy mi lesz, ha a kettőt összehozzuk? Harrisék megtették ezt és az eredmény pont azért izgalmas, mert kinézetében az így létrejövő kettős mutáns zebrahal (lat4anr21/+;kcnh2alof/+) eléggé hozza a repülőhalak “formáját”: elnyúlt mellúszókkal és felemás farokúszóval (felül rövid, alul hosszú) rendelkezik. Azaz két lépésben magunk is le tudtuk másolni a repülőhalak evolúciójának a talán leglátványosabb, thompsoni értelemben transzformációs lépését. (Természetesen mindez még nem elég a “repülőhalsághoz” és ezek a zebrahalak sosem fognak repülni. Ahhoz még számos más élettani és viselkedésbeli változásra lenne szükség.)
Bármennyire látványos is (és gumilapra kívánkozó) transzformáció, az “On Growth and Form”-ból sajnos pont a repülőhalak úszója hiányzik. De ez nem azért van, mert D’Arcy Thompsont esetleg nem érdekelték volna ezek a halak. A görög irodalomban nagyon is otthon levő Thompson maga fordította angolra Arisztotelész “Az állatok története” című művét, amiben a repülőhalak egyik első említése is található és ez kerülhetett be később, az “On Growth and Form” után három évtizeddel publikált “Glossary of Greek Fishes” című könyvbe is, amiben Thompson a klasszikus görög irodalomban említés szintén megjelenő halakat próbálta beazonosítani.
(A borítókép a Flickr-ről származik.)
Daane JM, Blum N, Lanni J, Boldt H, Iovine MK et al. (2021) Novel regulators of growth identified in the evolution of fin proportion in flying fish. bioRxiv 2021.03.05.434157; doi: 10.1101/2021.03.05.434157.
F.J. van Eeden, M. Granato, U. Schach, M. Brand, M. Furutani-Seiki, et al. (1996) Genetic analysis of fin formation in the zebrafish, Danio rerio. Development 123: 255-262.
Nüsslein-Volhard C. (2012) The zebrafish issue of Development. Development 139: 4099-4103 doi: 10.1242/dev.085217.
Perathoner S, Daane JM, Henrion U, Seebohm G, Higdon CW, Johnson SL, et al. (2014) Bioelectric Signaling Regulates Size in Zebrafish Fins. PLoS Genet 10(1): e1004080. doi: 10.1371/journal.pgen.1004080.
Stewart S, Le Bleu HK, Yette GA, Henner AL, Robbins AE, et al. (2021) longfin causes cis-ectopic expression of the kcnh2a ether-a-go-go K+ channel to autonomously prolong fin outgrowth. bioRxiv 790329; doi: 10.1101/790329.

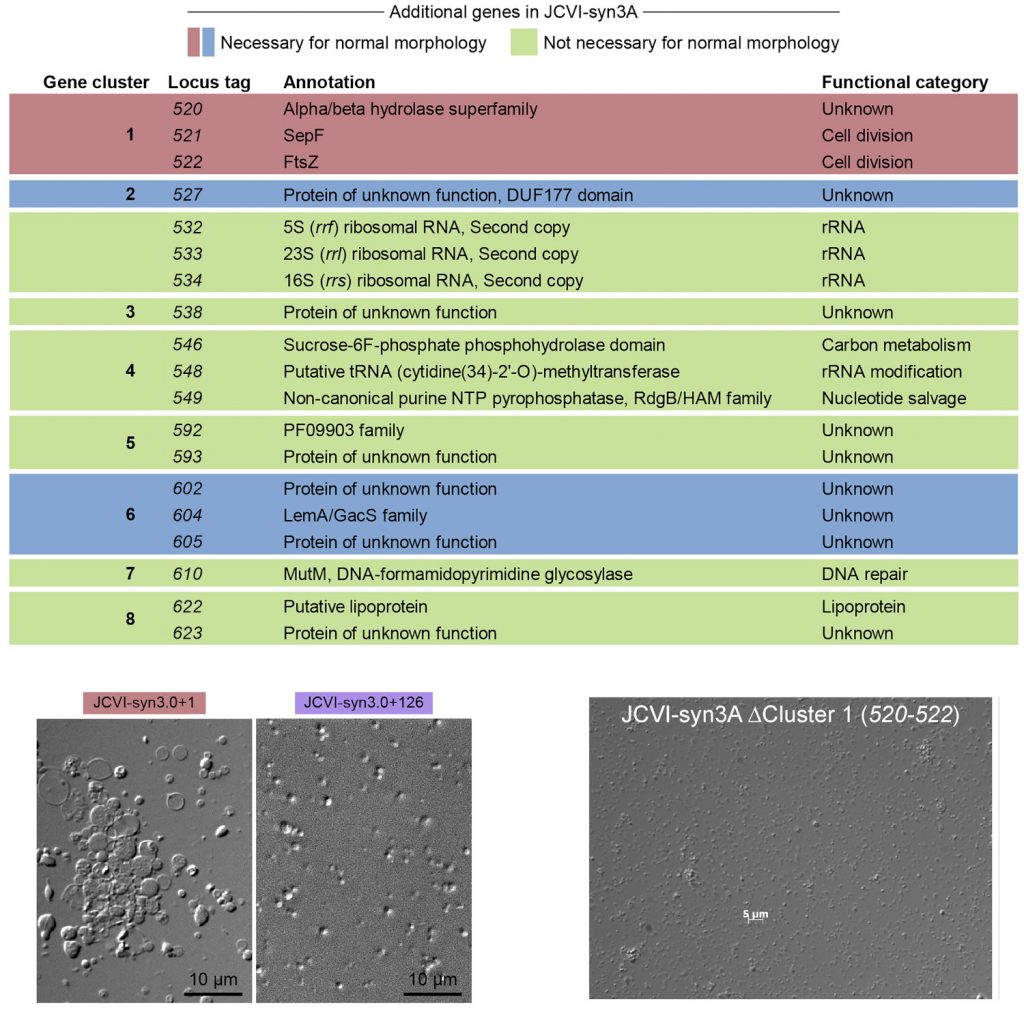
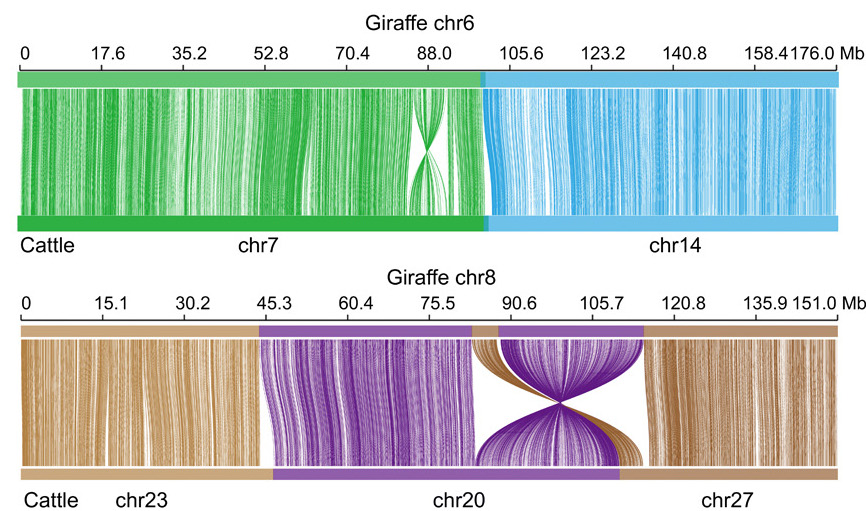

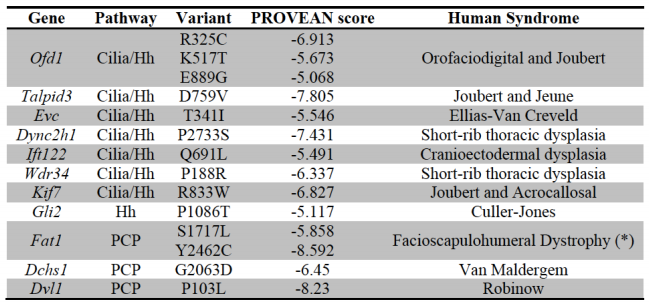
 Mennyire állandóak a genomok a vadonban? Milyen gyorsan változnak a gének? Egyáltalán hogyan közelíthetjük meg ezt a kérdést, találhatunk erre bármilyen választ? Bizony a biológiában akad olyan eset is, amikor a szemünk előtt alakult át gyökeresen egy vadon élő faj genomja, érdekes feleleveníteni ezt a lassan száz éves történetet.
Mennyire állandóak a genomok a vadonban? Milyen gyorsan változnak a gének? Egyáltalán hogyan közelíthetjük meg ezt a kérdést, találhatunk erre bármilyen választ? Bizony a biológiában akad olyan eset is, amikor a szemünk előtt alakult át gyökeresen egy vadon élő faj genomja, érdekes feleleveníteni ezt a lassan száz éves történetet.

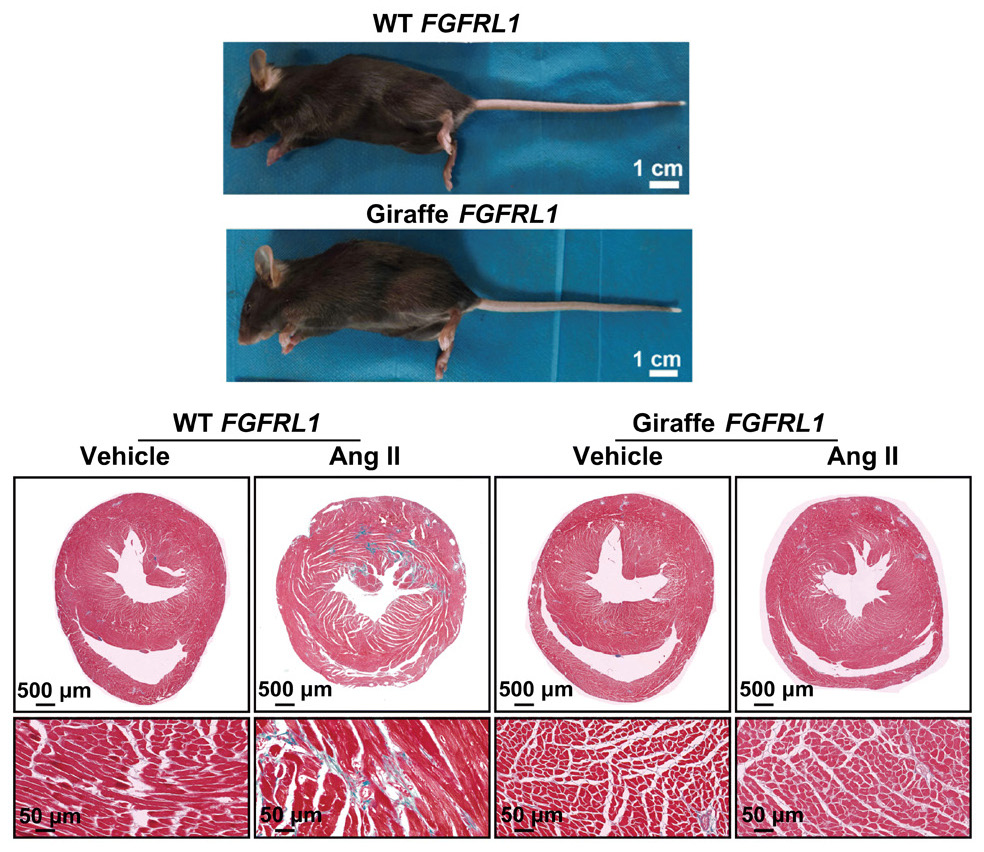
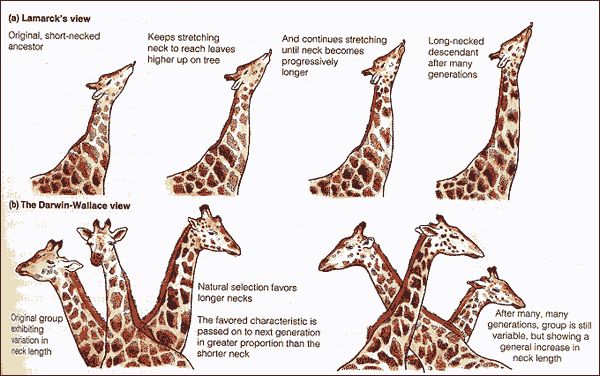 Ha van szó szerint tankönyvi példája a darwini evolúciónak, akkor az természetesen a zsiráfok kialakulása lenne. Hiszen ez az a folyamat, amivel leggyakrabban szemlélteni szokták a lamarcki és darwini evolúciós szemlélet közti különbségeket: míg a lamarcki nézet szerint a nyakát nyújtogató, okapi-szerű ős szorgalmas edzése vezetett a meghosszabbodott nyakhoz, addig a darwini nézet szerint a fajon belüli variancia azoknak az egyedeknek nyújtott szelekciós előnyt, akik hosszabb nyakkal rendelkeztek, ami hosszú generációk alatt az ő hosszú-nyakat biztosító génjeik elterjedését és így a faj/populáció általános nyakhosszabbodását okozta.
Ha van szó szerint tankönyvi példája a darwini evolúciónak, akkor az természetesen a zsiráfok kialakulása lenne. Hiszen ez az a folyamat, amivel leggyakrabban szemlélteni szokták a lamarcki és darwini evolúciós szemlélet közti különbségeket: míg a lamarcki nézet szerint a nyakát nyújtogató, okapi-szerű ős szorgalmas edzése vezetett a meghosszabbodott nyakhoz, addig a darwini nézet szerint a fajon belüli variancia azoknak az egyedeknek nyújtott szelekciós előnyt, akik hosszabb nyakkal rendelkeztek, ami hosszú generációk alatt az ő hosszú-nyakat biztosító génjeik elterjedését és így a faj/populáció általános nyakhosszabbodását okozta.
 Feltételezem, hogy mindenki evett már
Feltételezem, hogy mindenki evett már