Hány gén kell az élethez, vagy más szavakkal mi az a minimális genom, ami még lehetővé teszi, hogy egy sejt élni tudjon? Ez a viszonylag triviálisnak tűnő kérdés még mindig a legnehezebb kérdések egyike. Hiába szekvenáljuk és elemezzük most már ezrével a prokarióta élőlények genomjait, az utóbbi időben igazán nagy áttörést az elmúlt pár évben nem sikerült elérni. Ismerünk nagyon minimális genommal is fennmaradó parazita és szimbionta élőlényeket, de ezek gazdasejtjeiken kívül már nem életképesek, vagyis valamit elvesztettük abból a minimális génkészletből, ami az önálló élethez szükséges.
Az első emberi genom szekvenálásakor technológiai újításainak köszönhetően ismertté váló Craig Venter, pontosabban az általa alapított J. Craig Venter Institute (JCVI), most már több évtizede próbálja amolyan redukcionista eljárással meghatározni, hogy mi is lehet az a minimális genom, ami az élethez szükséges. Ez kb. azt jelenti, hogy fognak egy elég kis genomú baktériumot (esetükben a Mycobacterium mycoides-re esett a választás) és ebből próbálnak még kisebb genomú, de önállóan is életképes bakétriumot csinálni. Első próbálkozásuk inkább metodológiai érdekesség volt, ami azt bizonyította, hogy egy (ismert szekvenciájú) teljes genomot is meg lehet szintetizálni és aztán csak abból kiindulva is egy teljes értékű sejt jöhet létre.
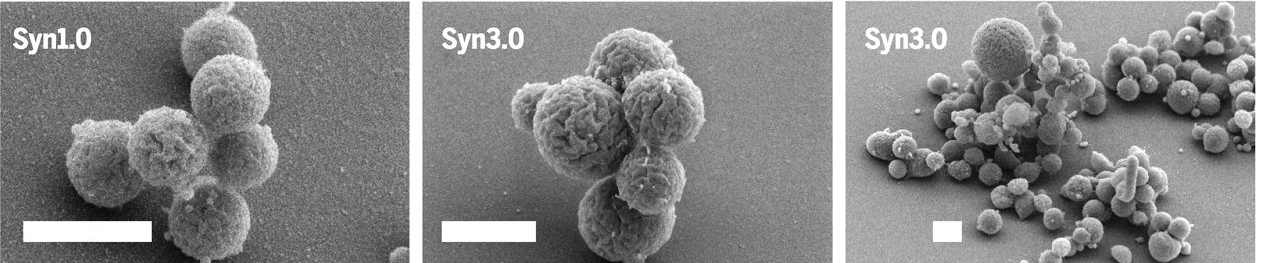
Fél évtizeddel később aztán bejelentették, hogy elkészült a JCVI-Syn3.0, ami már valóban fontos lépés volt a genom-minimalizálás felé, hiszen az M. mycoides genom eredeti 901 génjéből több százat kiszórtak és végül 473 maradt. A Sny3.0 kétségtelenül élt, de ahogy akkor is tudni lehetett, azért egy picit “magyar narancs” jellegű volt, osztódási nem mindig voltak szimmetrikusak, gyakran nagy vakuólák, máskor filamentózus szerkezetek jelentek meg a tenyészetekben.
Most ennek a furcsaságnak mentek utána a JCVI és az amerikai Nemzeti Standardok és Technológia Intézet (National Institute of Standards and Technology – NIST) kutatói. A munkában modern mikrofluidikai berendezéseket használtak a fura sejtmorfológiák pontos jellemzésére és a kísérlet alapját az képezte, hogy visszamentek megvizsgálni azokat a köztes állapotokat, amelyek a Sny3.0 létrehozásakor találtak – ti. akkor a Syn1.0 genomot nyolc részre osztották és az egyes részekből párhuzamosan vágták ki a nem esszenciális géneket, majd a nyolc “minimál” genomdarabot egyesítették a Syn3.0 genomjába.
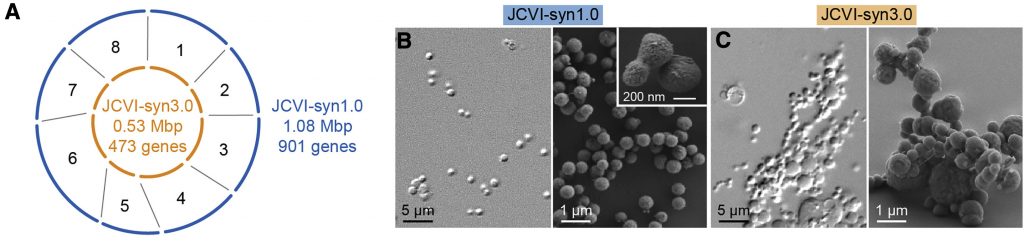
Most a kutatók először arra figyeltek fel, hogy a furcsán viselkedő fenotípus már akkor megjelenik, ha Syn1.0 genomban a 6. szegmenst helyettesítjük a minimál-szegmenssel (RGD6), illetve fordítva, ha a Syn3.0-ban a hatodik szegmenst egy nagyobbra cseréljük vissza, akkor megint normálisan viselkedő sejteket kapunk (ez lett a JCVI-Syn3A). (A Syn1.0 és Syn3.0 genomok közt a hatodik szegmensben 76 génnyi különbség van, de itt elég volt egy fragmenst visszapótolni, amiben 19 plussz gén van.)
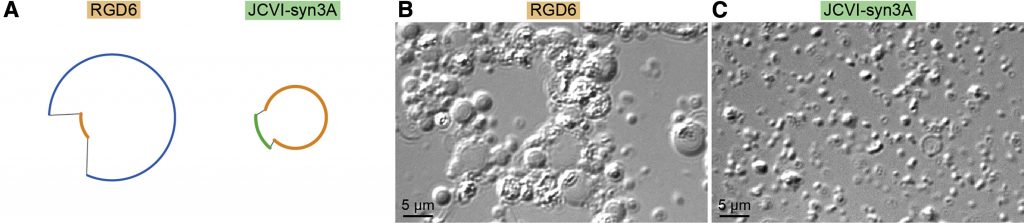
Vagyis adta magát, hogy a szóban forgó hatodik szegmenst, pontosabban az abból kitörölt géneket kell még részletesebben megnézni, hogy megérthessük, miért nem osztódik normálisan egy JCVI-Syn3.0 sejt.
Ahogy az alábbi táblázat is mutatja, összesen 19 gén különbözik a Syn3.0 és Syn3A genomok között, amelyeket nyolc további alszegmensbe (klaszterbe) lehetett osztani. Ezek közt voltak olyanok (pl. riboszomális RNS-eket kódoló gének), amelyek eredetileg is azért töröltek, mert redundánsak, vagyis több kópiában is jelen vannak a genomban, így nem volt meglepő, hogy ezek visszapótlása a Syn3.0 genomba nem okozott változást, a sejtosztódások továbbra is furák maradtak.
De volt pár másik, mint pl. az ftsZ, amelynek a fehérjeterméke (egy tubulin jellegű fehérje) korábban is ismerten fontos szerepet játszik a sejtosztódásban. Nem túl nagy meglepetésre, az ftsZ fontosnak bizonyult a normális morfológiához. Végül összesen 7 olyan gént azonosítottak, amelyek így vagy úgy, de együtt bepótolva a Syn3.0 genomba biztosították a normális osztódást.
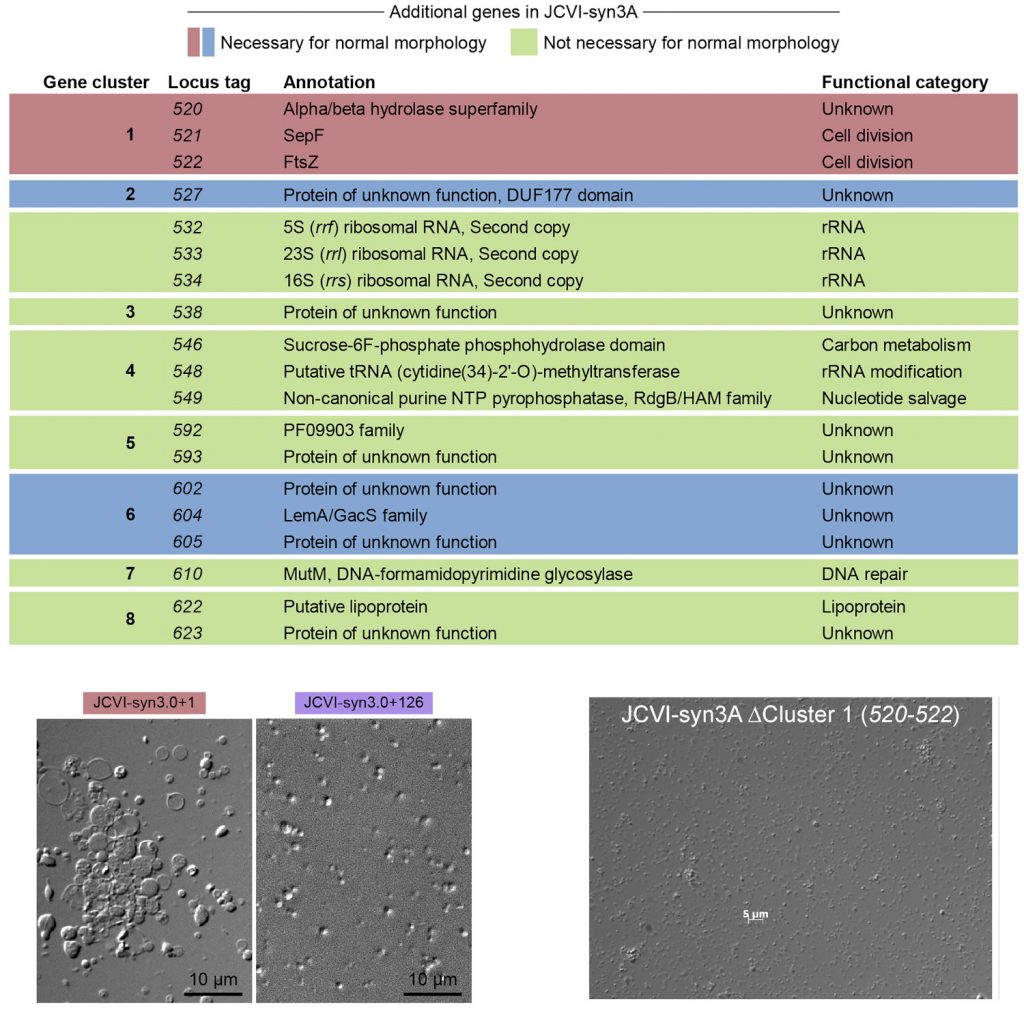
A történet itt véget is érne, de azért még van két érdekesség, amit mindenképpen megemlítenék: egyrészt, hogy ebből a mindössze 7 db. génből is öt olyan, aminek nem ismerjük az igazi funkcióját. És amikor egy tényleg alig pár száz génből álló genom esetében is egy ilyenbe belefutunk, akkor az mindig kellő perspektívába teszi, hogy a saját, sokkal komplexebb és közel húszezer fehérje-kódoló gént tartalmazó genomunkat mennyire érthetjük per pillanat.
A másik fontos dolog, hogy néha az egyszerű dolgok sem egyszerűek. Például bármennyire is fontosnak tűnik, de nem elegendő, ha csak az ftsZ-t tartalmazó klasztert visszük vissza a Syn3.0 genomjába, hanem a hét említett génre együtt van szükségünk a normális fenotípushoz. Ugyanakkor, ha a Syn3A genomból indulunk ki és abból töröljük ki az ftsZ-t és két további esszenciális(nak tűnő) gént tartalmazó klasztert, akkor a morfológia továbbra is normális marad. Vagyis a Syn3.0 genomon végzett “visszapótlási kísérletben” nem esszenciálisnak bizonyuló tizenkét génnek (köztük szintén van három ismeretlen funkcióval) valamiképpen kompenzálnia kell tudnia ennek a három génnek a hiányát. Hogy hogyan valósul meg ez a redundancia, azt még ötlet szintjén sem tudjuk.
És ez az, ahol a technológia tour de force, amire a JCVI kutatói mindig is hajlamosak, önmagában nem tud segíteni. Mert itt van ugyan egy minimális genom, de még mindig olyan alapvető kérdésekre is keressük a választ, hogy egyes esszenciális gének miért is annyira fontosak.
(A borítókép a J. Craig Venter Institute oldaláról származik.)
Pelletier JF, Sun L, Wise KS, Assad-Garcia N, Karas BJ, et al. (2021) Genetic requirements for cell division in a genomically minimal cell. Cell doi: 10.1016/j.cell.2021.03.008.


 A “miről írnék, ha épp lenne időm” sorozat újabb darabjához értünk, amit muszáj megemlítenem, még ha mélyebben nem is megyek most bele.
A “miről írnék, ha épp lenne időm” sorozat újabb darabjához értünk, amit muszáj megemlítenem, még ha mélyebben nem is megyek most bele.