Az állatvilágban (ha és amennyiben kétivarú fajokról beszélünk) a nemek kialakításáért felelős folyamatoknak (szexdetermináció) megdöbbentően széles spektrumával találjuk szembe magunkat. Csak a „magasabbrendű” magzatburkos gerincesekre szorítkozva eddig három egészen különböző logikájú rendszert írtak le: az emlősöknél és a madaraknál egyaránt az ivari kromoszómák játszák a fő szerepet, de míg előbbi esetben a hímek (XY), addig utóbbi esetben a nőstények (ZW) a különböző nemi kromoszómákat hordozó, ún. heterogametikus egyedek. A hüllők szerteágazó rokonságában szintén találkozhatunk kromoszomális szexdeterminációval, de a harmadik nagy kategóriát is a hüllők képviselői közt lelhetjük fel: sokuknál környezeti faktoroktól, pl. a fészekalj hőmérsékletétől függ a kifejlett példányok neme (nagyon sok hüllőfajt éppen ezért a klímaváltozás az ivararányok drasztikus eltolódása révén különösen érzékenyen érint). És persze előfordulhatnak előzőek furcsa kombinációi, mint a szakállas agámák esetén.
Adja magát a kérdés, hogy az egymással közelebbi, vagy távolabbi rokonságban álló magzatburkos csoportok milyen evolúciós utat járhattak be, ami végül az egyes csoportok eltérő ivarmeghatározó rendszereinek megjelenéséhez vezetett? Egy másik érdekes kérdés, hogy a sokféle szexdeterminációs rendszer közül melyik hasonlíthat legjobban a magzatburkosok utolsó közös ősében működő rendszerhez. A kérdés megvizsgálására a modern genetikai eszközök és új tudományágként az összehasonlító genomika kiváló lehetőségeket kínál.
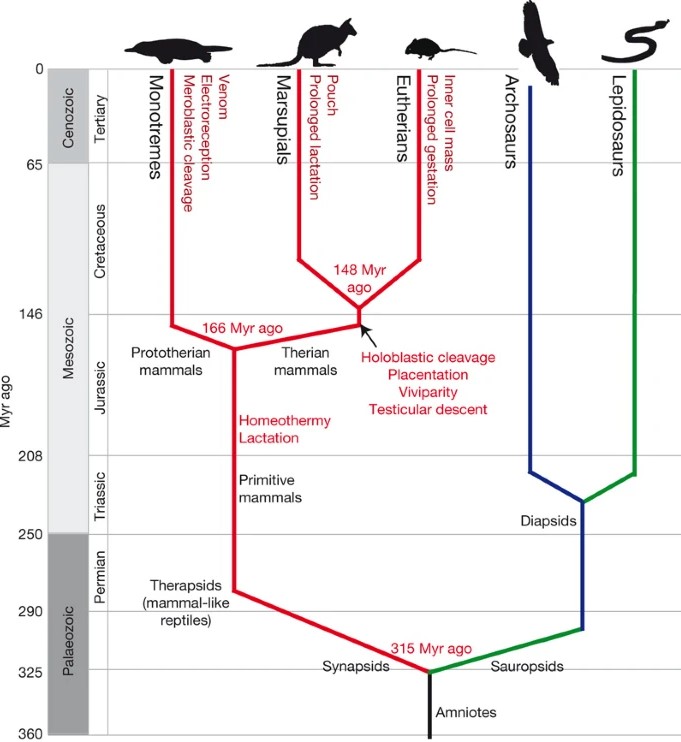
Vegyük rögtön (nem minden nárcizmustól mentesen) a saját példánkat! Ha kíváncsiak vagyunk rá, hogy honnan származik a Homo sapiens ivarmeghatározása, akkor először körbe kell nézni a méhlepényes emlősök (Placentalia) között, majd vetni kell egy pillantást az erszényesek (Marsupialia) szexkromoszómáira, végül meg kell nézni az emlősök osztályának legkorábban (kb. 160-180 millió éve) leágazott rendjét, a kacsacsőrű emlőst (Ornithorhynchus anatinus) és a 4 recens hangyászsünfajt magába foglaló Monotremata csoportot.
Ahogy az emlősök családfájának ágain ugrálva távolodunk saját fajunktól, úgy végigkövethetjük az X és az Y kromoszóma változásait is, de ha leérünk a fa legalsó ágához, ott nagy meglepetések fogadnak. A Monotremata csoportnak ugyanis nem pusztán 1, hanem rögtön 5 pár szexkromoszómája van. Ez az öt pár kromoszóma ráadásul egy nagyon különleges multikromoszómás komplexet alkot a sejtosztódások során, megelőzendő a homológok véletlenszerű kiosztását a leánysejtek között. Különösen fontos ez az ivarsejteket képző számfelező osztódás (meiózis) során, hisz egy ivarsejtbe (gaméta) vagy csak X, vagy csak Y kromoszómák kerülhetnek.
A Monotremata szexkromoszómák (miként más állatoknál is) tartalmaznak nem rekombinálódó, a nemek kialakítását szabályozó régiókat (SDR – sex determination region), illetve olyan régiókat, amelyek lehetővé teszik a kromoszómapárok egymásra találását – ezek a régiók ugyanúgy működnek, mint a nem-kromoszómák, azaz automszómák (nevük is ennen ered: pszeudoautoszómális régiók – PAR). A trükk, ami a különleges multikromoszómás komplex kialakulását eredményezi abban áll, hogy nem csak a kromoszómapárokon, de a sorban egymást követő X és Y kromoszómákon is találhatók egymáshoz illeszkedő PAR-ok. Így tehát a kacsacsőrű emlős X1-es kromoszómája nőstény egyedekben kapcsolódhat egy másik X1-hez és X2-höz is, míg hímekben az egyik X1 helyett Y1 áll. Az Y1 X2-höz, az X2 Y2-höz és X3-hoz, X3 Y3-hoz és X4-hez, ami Y4-hez és X5-höz, ami végül Y5-höz illeszkedik meióziskor.
További érdekesség, hogy a szekvenciaelemzések az így kialakuló kromoszómaláncok két végén is feltártak egy-egy, mostanra már erősen mutálódott PAR elemet, amelyek a mai Monotrematák őseinél elvileg lehetővé tehették, hogy a láncszerű multikromoszómás komplex gyűrűvé záródjon. Ha ez a hipotetikus őskromoszóma-komplex valóban képes volt a saját farkába harapni, az már csak azért is izgalmas, mert eddig az egész élővilágban csak egy növényél, pontosabban egy ligetszépe fajnál figyeltek meg gyűrűbe záródó meiotikus kromoszómaláncot. Ritka kuriózum hát az ilyen!
A kacsacsőrű emlős és a hangyászsün genomok feltérképezésének korai szakaszában, Rens és munkatársai közöltek egy izgalmas összehasonlító tanulmányt, amelyben akkor főleg in situ hibridizációs (FISH) módszerek segítségével vetették össze egymással a kacsacsőrű emlős és a hangyászsünök, valamint a méhlepényesek és a madarak ivari kromoszómáit. Ami már az elején feltűnő volt, hogy a Monotremata fajok szexkromoszómái néhány apróbb különbséget leszámítva erősen konzerváltak. Az egy már korábban megfigyelt különbség, hogy a hangyászsünök Y5-ös kromoszómája hiányzik. Ez a kromoszóma a kacsacsőrű emlősnél is meglehetősen apró, így korábban elfogadható magyarázatnak számított, hogy a hangyászsünök egyszerűen elveszítették azt valamikor az evolúciójuk során. Rensék kutatásai annyival árnyalták ezt a képet, hogy a hangyászsün Y3-as kromoszómáján találtak olyan régiókat, amelyek az elveszettnek hitt Y5-nek felelnek meg. Az újabb értelmezés szerint tehát ez a kromoszóma nem eltűnt, csak integrálódott a hármas sorszámú Y kromoszómába, hasonlóan ahhoz, amikor két kártyapaklit egybekeverünk.

Visszatérve eredeti kérdésünkhöz, hogy honnét származik a méhlepényesek, illetve az ember X és Y kromoszómája, egy dolgot már biztosan elmondhatunk: nem a legelső emlősöktől örököltük őket. A Monotremata szexkromoszómák ugyanis nem mutatnak semmilyen homológiát a Placentalia szexkromoszómákkal. A mi magányos és aprócska Y kromoszómánkon található SRY lókusz pedig, ami a méhlepényeseknél kulcsszerepet tölt be a herék fejlődésének kezdetén, s így végső soron a hím nemi jellegek megjelenésének forrása, mind a kacsacsőrű emlős, mind pedig a hangyászsünök genomjából hiányzik.
A méhlepényesek X kromoszómájának hosszú karján található egy, a dóziskompenzációban fontos lókusz, az XIST. Ennek a szakasznak a megfelelőjét kimutatták ugyan a kacsacsőrű emlős genomjában, de nem az egyik ivari kromoszómán, hanem a 6-os autoszómán, arra pedig egyelőre semmi nem utal, hogy ez a lókusz bármiféle szerepet is betöltene a kacsacsőrű emlősök szexdeterminációjában. Jelenleg úgy tűnik tehát, hogy a Monotremata és a többi emlős szexdeterminációs rendszere egymástól függetlenül jelent meg a két csoport elválását követően, és semmiféle közös eredettel nem bír.
Nagyfokú homológia mutatható ki azonban a kacsacsőrű emlős X5-ös, a hangyászsünök X4-es szexkromoszómája, valamint a madarak Z kromoszómája között. A madarak szexdeterminációjában fontos szerepet játszó, dózisfüggő transzkripciós faktorok, a DMRT-1, -2 és -3 mind megtalálható a kacsacsőrű emlős X5-ös szexkromoszómáján, de találtak olyan Z kromoszómára térképezett géneket is, amelyek ortológjai a kacsacsőrű emlős X2 és X3 ivari kromoszómáján helyezkednek el. Ugyanezek a Monotremata kromoszómarégiók nagyfokú egyezést mutattak a humán 9-es autoszómával is, ami többé-kevésbé megfeleltethető a madarak Z kromoszómájának, illetve a humán 5-ös és 18-as kromoszómáknak azon részleteivel, amelyek a madarak 2-es autoszómájával hibridizáltathatók.
Azt jelentené ez, hogy a Monotremata és a madár szexdetermináció (az XY és a ZW rendszerek eltérő logikája ellenére) azonos gyökerekre vezetnek vissza? A hüllőket és madarakat magába foglaló Sauropsida, valamint az összes mai emlőst, és egy seregnyi emlősszerű őshüllőt tartalmazó Synapsida csoportok több mint 300 millió éve, a késő Karbonban válhattak el egymástól. Ha tehát elfogadjuk az iménti felvetést, azzal azt is feltételezzük, hogy a legkorábbi magzatburkosok nemmeghatározása szempontjából a DMRT gének fontosak lehettek.
Árnyalja a képet egy idén januárban a Nature hasábjain megjelent cikk, amelynek szerzői már elég részletes genomi információk birtokában kísérelnek meg rendet tenni az emlős kromoszómák háza táján. A kísérlet nem volt sikeres, hisz a rendcsinálás közben a kupleráj, amelybe a fenti sorok olvasása közben betekintést nyerhettünk, csak sokszorosára duzzadt.
A fali gyík, a házityúk, a kacsacsőrű emlős, a tasmán ördög, egy oposszum faj és az ember genomjának konzervált kromoszómális régióit és e régiók egymáshoz viszonyított pozícióját elemezték, s megállapították, hogy e régiók igencsak össze lettek keverve az elröppent év-százmilliók alatt. A szerzők ötszáznál is több független kromoszómaátrendeződési eseményt azonosítottak (transzlokációk, kromoszómafúziók és fragmentálódások).
Most képzeljük el, hogy valaki egyszer az aztalon felejtett néhány pakli kártyát, amiket minden arra járó ember keverget egy kicsit. Nekünk pedig egyszer csak a mániánkká válik, hogy lépésről-lépésre megértsük a paklik összekeveredésének történetét. Szerencsénkre a kártyaasztal a rajta lévő paklikkal együtt, valami rejtélyes módon párszor lemásolta saját magát, így az egyes másolatokból kinyerhetünk némi információt arra vonatkozóan, hogy a másolatkészítés pillanatában hogyan néztek ki az „ősasztal” „őskártyapaklijai”. De jaj! Valaki aljas módon tovább keverte az összes másolat-kártyapaklit is! Ön hogy állna neki az eredeti kártyapaklik rekonstrukciójának?
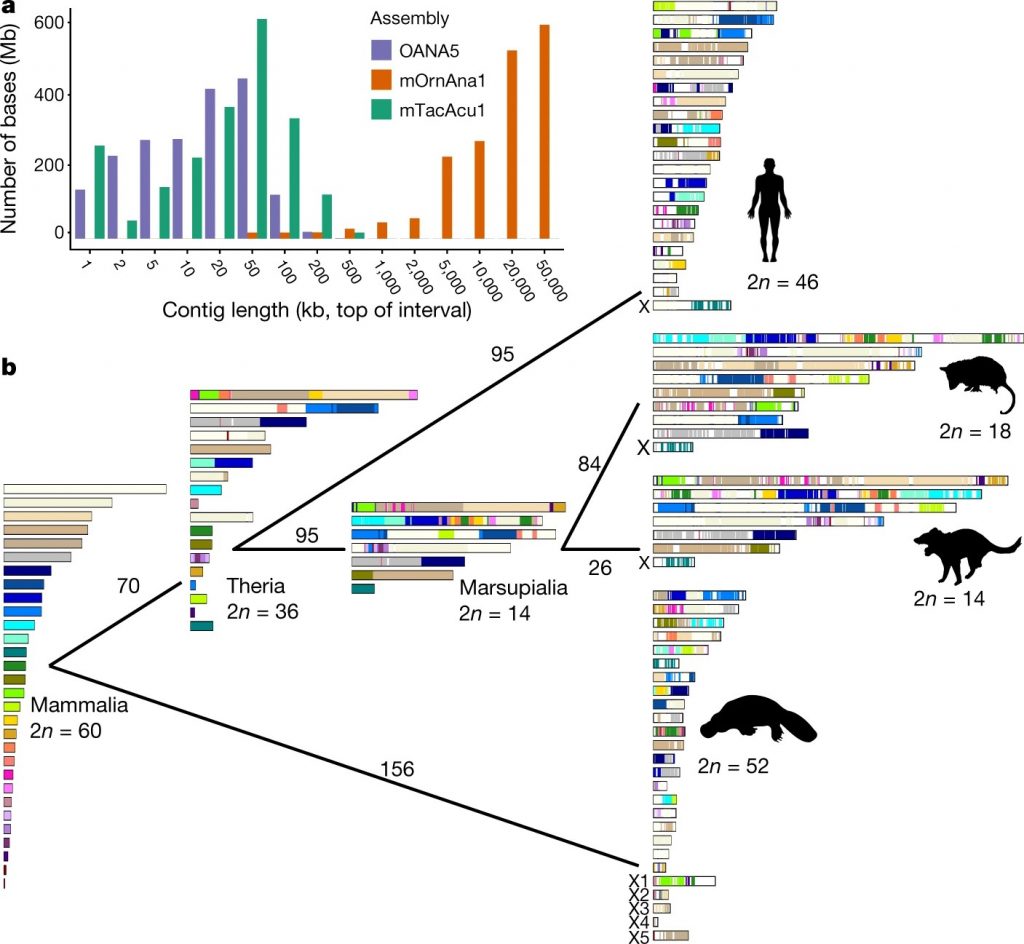
A megoldás kulcsát az asztalmásolatok jelentik. Ha ezeket összehasonlítva az egyes paklik kártyasorrendjében hasonlóságot találunk, akkor az a másolatkészítés előtt alakult ki, ha pedig különbségeket, akkor azok olyan kevergetésből származnak, amelyek a másolatkészítést követően történtek. A kártyás allegóriát feloldva: a kártyalapok kromoszómarégiókat, a paklik kromoszómákat, az asztalok pedig fajokat jelölnek, amelyek összehasonlítása révén nyílik lehetőségünk a keverés történetének és végső soron az „őskártyapaklik” rekonstrukciójára. Zhou és munkatársai elvégezték hát ezt a heroikus rekonstrukciót, és egyebek mellett megállapították, hogy az emlősök közös ősének valószínűleg 30 pár kromoszómája volt. A szerzők azon túl, hogy mind a 30 őskromoszómáról (Mammalian Ancestral Chromosome – MAC) leírták a konzervált régiók relatív sorrendjét, fontos megállapításokat tettek a szexkromoszómákkal kapcsolatban is.
Úgy tűnik, hogy a Monotremata 5 pár ivari kromoszómájának mindegyike több, különböző kártyapakli lapjaiból tevődött össze. Az a hasonlóság tehát, amit anno Rens és szerzőtársai a madarak és a Monotremata szexkromoszómák között megfigyeltek szinte biztosan nem arra vezethető vissza, hogy a madár Z és a kacsacsőrű emlős X5 kromoszómája egy közös őskromoszóma két távoli leszármazottja lenne.
Az új tanulmányok fényében az a fentebb megfogalmazott elmélet is megingott kissé, hogy DMRT gének ivari determinációban betöltött szerepe egy ősi magzatburkos vonás lenne. Hiszen egy ilyen viharos sorsú kártyaasztalnál nem elképzelhetetlen az sem, hogy két független kiosztásban kap egy gén hasonló szerepet, s kerül mondjuk szexkromoszómára. Tartsuk szem előtt, hogy a madarak és az emlősök utolsó közös őse több mint 300 millió éve élt a Földön, és hogy ismerünk olyan állatokat is, ahol egy fajon belül kettő, vagy több tök különböző szexdeterminációs rendszer is működik!
Na és mit tudunk meg végre a saját ivari kromoszómáinkról?
Korábban említettük az Y kromoszómánkon található SRY lókuszt, amely a Theria (tehát erszényes és méhlepényes) közös ősben már jelen volt és a maihoz hasonló szerepet tölthetett be, de a Monotremataban még nyomát sem látni. Az X kromoszómánk szintén a Monotremata-Theria elvállást követően jelenhetett meg, de a méhlepényeseknél fontos dóziskompenzációs gén, az XIST csak a méhlepényes-erszényes elvállást követően bukkant fel az X kromoszóma történetében. Fontos különbség még a kenguruk és az ember X kromoszómája között, hogy a méhlepényesek X kromoszómája az ősidőkben fuzionált egy ősi Theria autoszómával, míg az erszényeseknél ilyen fúziós esemény nem történt.
Szövevényes történetek játszódhattak a kártyaasztalok körül, és egyenlőre csak a legfontosabb események vázlatát látjuk. Ugyanakkor nem tudunk semmit azokról a játékosokról, akiknek a lapjárása olyan szerencsétlenül alakult, hogy kihajították őket az evolúció kaszinójából, és azok se buktak még le, akik cinkelt kártyával játszottak. Egyszer majd talán ezekre a történetekre is fény derül.
__________
Nicole Valenzuela és mtsai. (2019) Extreme thermal fluctuations from climate change unexpectedly accelerate demographic collapse of vertebrates with temperature-dependent sex determination” Scientific Reports
Craig A Smith és mtsai. (2009) The avian Z-linked gene DMRT1 is required for male sex determination in the chicken Nature
Hieronim Golczyk és mtsai. (2014) Translocations of Chromosome End-Segments and Facultative Heterochromatin Promote Meiotic Ring Formation in Evening Primroses. Plant Cell
Willem Rens és mtsai. (2007) The multiple sex chromosomes of platypus and echidna are not completely identical and several share homology with the avian Z. Genome Biology
Yang Zhou és mtsai. (2021) Platypus and echidna genomes reveal mammalian biology and evolution. Nature

Bocs, lehet, hogy bennem van a hiba, ezt nem tudtam kovetni, pedig altalaban nagyon elvezem az irasaitokat.
Biztosan nem.
Valószínűleg én bonyolítottam túl. Bocsánat.
Röviden összegezve arról van szó, hogy az emlősöknél jelenleg megfigyelhető kromoszómák egy ősi kromoszómaszett feldarabolódásával és a fragmentumok véletlenszerű egybeolvadásával jött létre. A kacsacsőrű emlősnek és nekünk hiába van ugyan úgy XY alapú szexdeterminációnk, ezek a kromoszómák eltérő eredettel rendelkeznek. Ami pedig ennél egy fokkal meglepőbb, hogy a kacsacsőrűeké a madarak ivari kromoszómáival mutatnak némi hasonlóságot.
Hogy miből mennyi van és hogyan alkotnak a kromoszómák komplexeket, az már csak hab a tortán.