A genetikai anyag változatosságának kialakításában az egyszerű mutációkon, kormoszóma átrendeződésken és genom duplikációkon túl fontos szerepet játszanak az ún. “ugráló gének”, a transzpozonok is [1]. Ezek olyan DNS szekvenciák, amelyek képesek a genom különböző helyeire beszúródni (vagy miután kivágódtak előző “lakhelyükről”, vagy anélkül, másolataik révén) többé-kevésbé (általában kevésbé) véletlenszerűen. (E tulajdonságuk miatt szeretik őket “önző DNS elemeknek” becézni, bár Richard Dawkins “önző gén” definíciója azért egy kicsit másra vonatkozik.) Egy (retro)transzpozon ugrálásának egy sor különböző következménye lehet, egy-egy gén működésképtelenné tételétől kezdve (ez a gyakoribb), új gének, exonok, promoterek és szabályozó szekvenciák létrehozásán át, kromoszomális átrendeződések okozásáig.
S mivel a gerinces genomok nagy számban tartalmaznak transzpozonokat (pl. az emberi genomnak közel fele ilyen), még viszonylag alacsony aktivitás mellett sem elhanyagolható működésük következménye. Ez a következmény lehet káros, lásd a kutyákban viszonylag gyakori narkolepszia is egy túl aktív transzpozon eredménye, de evolúciós mértékben lehet igen fontos is: például a patogénekkel való folytonos harc szempontjából nagyon fontos adaptív immunrendszerünk egyik kulcsgéne, a RAG1 rekombináz transzpozon eredetű [2]. Ha a transzpozon ugrásának eredménye valamilyen módon csökkenti az élőlény fittneszét, akkor a természetes szelekció hamarosan kiszórja az új változást, ellenben, ha az ugrás hatástalan, akkor a változás akár meg is maradhat (ha pedig kifejezetten hasznos, akkor jó eséllyel rögződik) a genomban. Egy-egy ilyen rögzült transzpozon jelenléte ill. hiánya, hasonlóan egyes mutációkhoz árulkodó lehet a törzsfejlődési kapcsolatokra nézve is: hiszen ha az ugrás két csoport szétválása után/során következik be egyikükben, akkor az abba a társaságba tartozó összes faj hordozni fogja az új elemet, míg a másik csoportba tartozók pedig nem. Ezt a logikát felhasználva támasztották alá a közelmúltban azt az elméletet, hogy a vendégízületesek (Xenarthra) a méhlepényes emlősök legősibb csoportját alkotják [3].
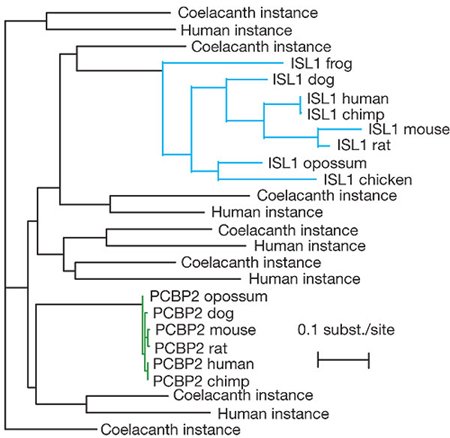
Az egyes fajokban jelen levő ugráló gének szekvenciájának ismeretében nyomozni is lehet: azt keresni, milyen gének szabályozó szekvenciák eredhetnek transzpozonokból. A héten két cikk jelent meg [4,5] ahol ilyesmit keresgélnek. Az első cikk [4] szerzői egy olyan transzpozont fedeztek fel, amely mintegy 410 millió éve jelent meg a ma élő bojtosúszóshalak és négylábú gerincesek közös ősében. Az LF-SINE-nak keresztelt elem nem lelhető fel a sugárúszóshalak és primitív gerinchúrosok genomjában, de az összes “fejlettebb” gerinces DNS állományában ott figyel. És nem nem lehet arra sem panaszkodni, hogy kizárólag potyázott volna: az emlős genomban például mindjárt két olyan LF-SINE eredetű szekvenciát leltek, amelyeknek fontos szerepük van: az mRNS feldolgozásáért felelős PCBP2 gén egyik exonja minden jel szerint LF-SINE eredetű, és ami még érdekesebb, a mozgást irányító motor neuronok fejlődésében kulcsfontosságú isl1 gén expresszióját szabályozó ún. enhancer régió is így jött létre.
A klasszikus bojtosúszós hal, a Latimeria menadoensis genomjából még csak kis részeket szekvenáltak meg, de ennek alapján úgy tűnik, hogy szárazföldi rokonaikkal ellentétben az LF-SINE működése még mindig aktív. Míg előbbiek genomjaiban pár száz körül van a kópiaszáma, a teljes Latimeria genomban 105-re saccolják.
A második szóbanforgó tanulmány szerzői egy fiatalabb transzpozíciót azonosítottak. Ez “mindössze” 48-60 millió éve következett be a főemlősök ősében, ahol a Hsmar1 nevű transzpozon ugrott a metiltranszferáz aktivitású SET gén mögé, ahol átalakult egy immár új gén, a SETMAR, exonnává. A SETMAR feladata még nem tisztázott, de mivel a főemlősökön belül kevesett változott a szekvenciája, gyanítható, hogy valami lényeges funkciója van.
[1] Kazazian HH Jr. (2004) Mobile elements: drivers of genome evolution. Science 303: 1626-1632.
[2] Kapitonov VV, Jurka J. (2005) RAG1 core and V(D)J recombination signal sequences were derived from Transib transposons. PLoS Biol. 3(6): e181.
[3] Kriegs JO, Churakov G, Kiefmann M, Jordan U, Brosius J, Schmitz J. (2006) Retroposed elements as archives for the evolutionary history of placental mammals. PLoS Biol. 4(4): e91.
[4] Bejerano G, Lowe CB, Ahituv N, King B, Siepel A, Salama SR, Rubin EM, Kent WJ, Haussler D. (2006) A distal enhancer and an ultraconserved exon are derived from a novel retroposon. Nature 441: 87-90.
[5] Cordaux, R, Udit, S, Batzer, MA and Feschotte, C (2006) Birth of a chimeric primate gene by capture of the transposase gene from a mobile element. PNAS 10.1073/pnas.0601161103