
Most, hogy a többség már (talán) megbarátkozott a PCR-alapú vírosdiagnosztika mibenlétével, ideje szemügyre vegyük, mit tartogat a jövő.
A nagyobb járványok kirobbanásában szerepet játszó patogének mindig nagy figyelmet kapnak a biológiai kutatások terén is, mivel ez igen közvetlenül érinti a társadalmunkat. A molekuláris diagnosztikai technikák fejlődésével az utóbbi évtizedek folyamán a fertőző betegségek diagnosztizálása, különösen a vírusdiagnosztika, rendkívüli gyorsasággal fejlődésnek indult. A vírustesztek legújabb generációja az ún. CRISPR-rendszeren alapul – ezeknek a működési elvét igyekszik a poszt alább összefoglalni, kitérve arra, hogy ezt hogyan lehet specifikusan a SARS-CoV-2 vírus detektálására használni. (Ugyanakkor, fontos hozzá tenni, hogy ellentétben a hosszú ideje és széles körben használt, PCR-alapú diagnosztikával, az ilyen, CRISPR-alapú rendszerek csak nemrég kaptak először engedélyt klinikai felhasználásra.)
A CRISPR rendszer
Napjainkban a legprecízebbnek kikiáltott géntechnológiai módszer a CRISPR-alapú genomszerkesztés, mely lehetővé teszi az örökítőanyag szekvencia-specifikus megváltoztatását. Ez már önmagában is figyelemreméltó lenne, de az utóbbi években egyre-másra jelentek meg ennek a sokrétű molekuláris rendszerek, amelyek különböző természetes CRISPR rendszereket igyekeznek egyéb célokra, például vírusdiagnosztikára is használni.
A CRISPR alapelve a sejtek idegen DNS (vírus vagy plazmid) ellen kialakított védelmi rendszerén alapszik. A legtöbbet emlegetett és használt CRISPR rendszert a Streptococcus pyogenes nevű baktériumból származik és lényegében a sejt egyfajta önvédelmi mechanizmusaként működik, ahol egyes Cas (CRISPR associated genes) fehérjék felismerik és feldarabolják az idegen DNS-t (ami pl. a baktériumot megtámadó fágból származhat), majd egy fajra jellemző “kitöltő” (spacer) szekvencia mögé építik be a genomi DNS-be. Ahogy ez a jelenség ismétlődik fokozatosan kialakul egy olyan rész a genomban, ahol az ismétlődő “kitöltő” szekvenciák közt a korábbi fág-támadások “szekvencia-emléknyoma” lelehető fel. Az így keletkező szekvenciát hívják CRISPR-nek (clustered regularly interspaced short palindromic repeats). Ezen szekvencia alapján keletkeznek a sejtben azok a rövid, irányító gRNS (guide RNS) molekulák, amelyek segítségével a sejt felismeri, ha újból ugyanannak a fágtámadásnak az alanyává válik. A gRNS szekvenciája ugyanis megfeleltethető lesz a fág DNS-ének, és ezt felhasználva a DNS hasító (endonukleáz) aktivitással rendelkező további Cas fehérjék (a S. pyogenes esetében a Cas9) elhasítja az idegen DNS-t, ezzel is gátolva a fertőzést. (Josiane E. et al 2010)
A modern géntechonológia ezt a rendszert használja ki, pontosabban az, hogy az endonukleáz aktivitással rendelkező Cas fehérjék a megfelelő gRNS-ek segítségével bármelyik fajban (és nem csak baktériumokban, hanem eukariótákban is), elvileg tetszőleges DNS-t szekvenciaspecifikusan tudnak hasítani. Ez a rendszer egyszerűbb, könnyebben és pontosabban megtervezhető, az eddig használt genomszerkesztési módszereknél , ezért is tudott gyorsan elterjedni (Haifeng W. et al, 2016).
CRISPR-Cas rendszer felhasználása a vírusdiagnosztikában.
A S. pyogenes Cas9-alapú CRISPR-rendszerére épülő rendszerek azonban csak egy szeletét jelentik azoknak az új kezelési eljárásoknak, amelyek a közeljövőben mindennapjaink részeivé válhatnak. Mivel ez a rendszer DNS hasításra “szakosodott”, klinikai alkalmazásánál is elsősorban olyan retrovírusok (pl. HIV) jöhetnek célba, amelyek a saját genetikai anyagukat a sejtek genomjába másolják bele. Ilyenkor a Cas9 fehérjét a vírus DNS-ét célzó gRNS-ekkel juttatják be a fertőzött sejtekbe, ami elősegít a vírus inaktiválását (Haifeng W. et al, 2016).
Azonban további, egyéb baktériumokban leírt CRISPR-rendszerek (és az azokhoz tartozó Cas fehérjék) bevonásával olyan vírusdetektálásra alkalmas gyorsteszteket is létrehoztak, amelyek gyökeresen átalakíthatják az epidemiológiai megfigyelési rendszereket (Kostyusheva, A., et al, 2020). A leginkább bevált technikák, a SHERLOCKv2 (specific high sensitivity enzymatic reporter unlocking version 2) és DETECTR (DNA endonuclease targeted CRISPR trans reporter) fantázianévre hallgatók, amelyek lehetővé teszik a patogén nukleinsavak gyors kimutatását rendkívül kis mennyiségében is (Kocak&Gersbach, 2018). Előbbi a Cas13, utóbbi pedig a Cas12 endonukleázok aktivitásának kihasználásán alapul.
A DETECTR rendszer
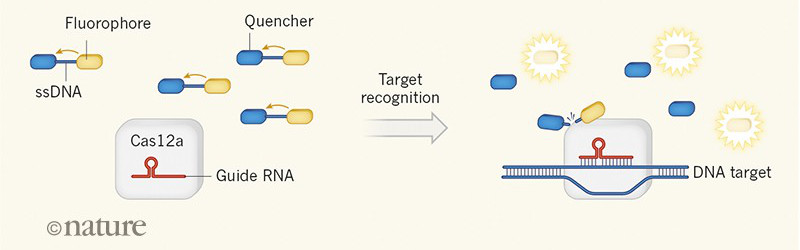
A CRISPR-Cas családon belül a Cas12 egy RNS-vezérelt DNáz, amelyben a cél-felismerés után egy aspecifikus egyszálú DNS (ssDNS) hasító képesség jelenik meg. Ezt kihasználva tervezhetők olyan ssDNS-riporterek, amelyek (aspecikus) hasítása vagy egy érzékeny detektor segítségével észlelhető (pl. fluoreszcencia megjelenése) vagy látható jelet hoz létre egy papírszalagon a laterális áramlásos (lateral-flow) módszer használatával (hasonlóan a már ismertebb ellenanyag gyorstesztekhez) (Chen és mtsai., 2018).
Az eddigi tesztelések során a CRISPR-Cas12 alapú kimutatási eljárást egy olyan LOD (limit of detection) érték jellemzi, amely alacsonyabb, mint ami vírus jelenlegi klinikai mintákban történő kimutatásához szükséges minimális szint. A CRISPR-diagnosztikai módszer itt bemutatott fő előnye a hordozhatóság és az alacsony költségek (1-2 USD / reakció).
A SHERLOCK rendszer
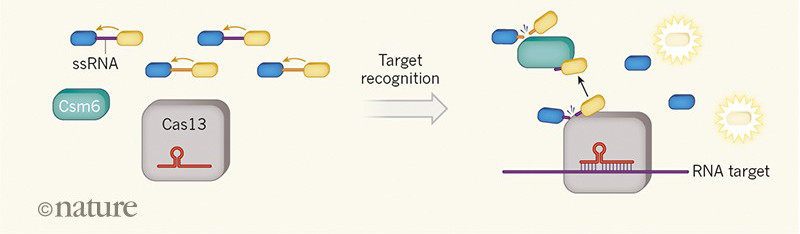
A SHERLOCK több CRISPR-rendszerhez is adoptálható (így a Cas12-höz is), de először a Cas13 kapcsán írták le (Gootenberg és mtsai., 2017). Utóbbi egy RNS-t felismerő Cas nukleáz, amelynél a cél megkötése szintén indukál egy aspecikfikus (kollaterális) nukleáz aktivitást, aminek – ebben az esetben – a környező RNS-ek látják kárát. Ennek megfelelően – ebben a rendszerben – a riporter a DETECTR-hoz hasonló logikájú, de RNS-alapú molekula kell legyen.
Bár látszólag a klasszikus SHERLOCK-rendszer előnye lehetne az RNS vírusok (például koronavírusok) esetében, hogy közvetlenül az RNS-t képes kimutatni, a valóságban az érzékenység megnövelése miatt szükség van a mintákban levő nukleinsavak sokszorosítására, ami általában DNS-alapú folyamat. Így, a Cas13-alapú SHERLOCK esetében szükség van egy köztes lépésre, amikor a felsokszorozott DNS-t visszaírják RNS-é (IVT – in vitro transzkripció). Ez pedig kicsit fölösleges lépés, ezért is tértek át a módszer kifejlesztői szintén a Cas12-alapú detektálásra (Gootenberg és mtsai., 2018).
Koronavírus detektálása CRISPR használatával
A SARS-CoV-2-nél tapasztalt rendkívül gyors világszintű terjedés sebességének csökkentésére, a vírus elleni hatékony védekezés érdekében fontos, hogy a fertőzést minél gyorsabban, lehetőleg minden fertőzött esetében és minél koraibb fázisban ki tudjuk mutatni. A CRISPR-Cas rendszerek a szekvenciaspecifitásuk következtében megfelelőek lehetnek ilyen diagnosztikai célokra, ráadásul két fontos előnyük van a jelenlegi detektálási rendszerekkel szemben: gyorsabbak és lényegesen olcsóbbak (ugyanakkor, ahogy már említésre került, egyelőre egyetlen teszt-kit kapta meg a megfelelő klinikai minősítést).
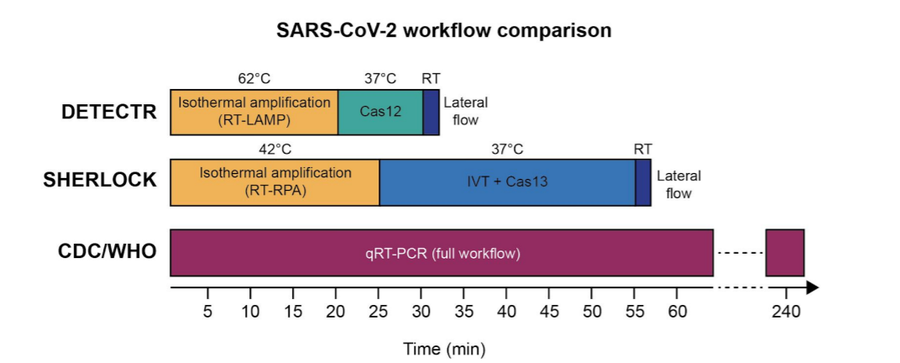
A korábbiak alapján nem volt kétséges, hogy mind a DETECTR, mind a Cas12-alapú SHERLOCK rendszerek alkalmasak lehetne a SARS-CoV-2 detektálására, így már februárban megindult a tesztek fejlesztése és azóta is folyamatos. Hogy minél gyorsabb és olcsóbb legyen a detektálás a legtöbb aktuálisan fejlesztett teszt nem fluoreszcens-alapú, hanem a klasszikusabb laterális áramlás technológiát használja és az eredményeket egy egyszerű színreakció jelzi ki.
Az érzékenység növelése miatt (hiszen van, amikor csak néhány vírus partikulum lesz az orrgarat-mintákban), a mintaelőkészítés a vírusgenom sokszorosításával kell induljon. Ehhez először a virális RNS genomból reverz transzkripció révén DNS-t készítenek és ezt követi a sokszorosító láncreakció egy különleges formája, ami a hőmérséklet folyamatos változtatása nélkül is működik, az ún. hurok-mediált isotermális amplifikáció (loop-mediated isothermal amplification – LAMP). Ez a reakció állandó (60–65 °C) hőmérsékleten lezajlik, speciális DNS-polimeráz segítségével, így nincs szükség a hagyományos PCR-gépre, csak egy egyszerű vízfürdőre, vagy szobai melegítőre (Notomi, et al., 2000).
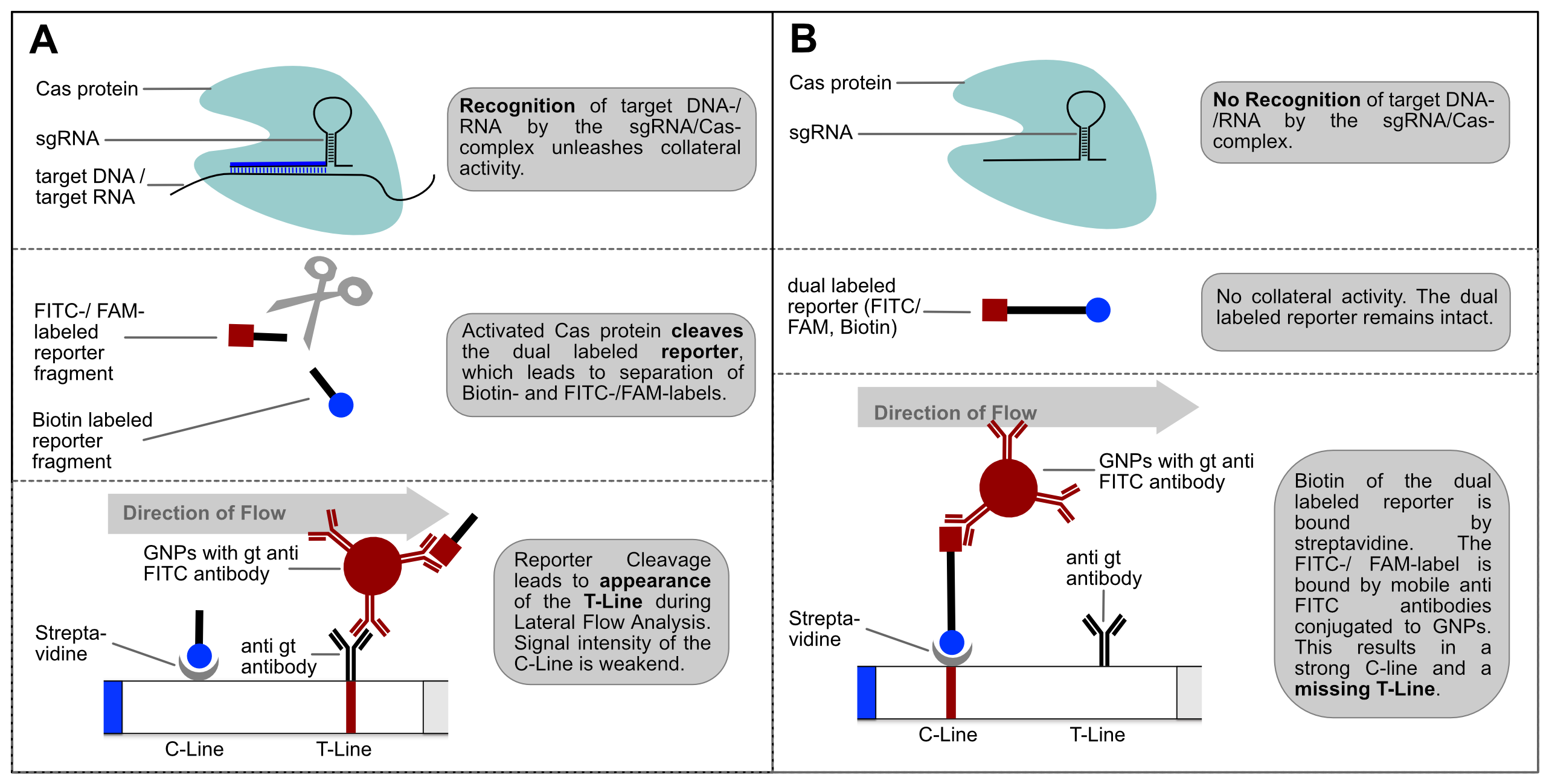
Amennyiben a levett mintában jelen volt a vírus, a koronavírus szekvenciájra tervezett gRNS segítségével a Cas12 észleli az egyezést és elhasít egy egyszerű szerkezetű ssDNS riporter molekulát, ezzel jelezve a fertőzést (Broughton et al., 2020; Curti, et al., 2020).
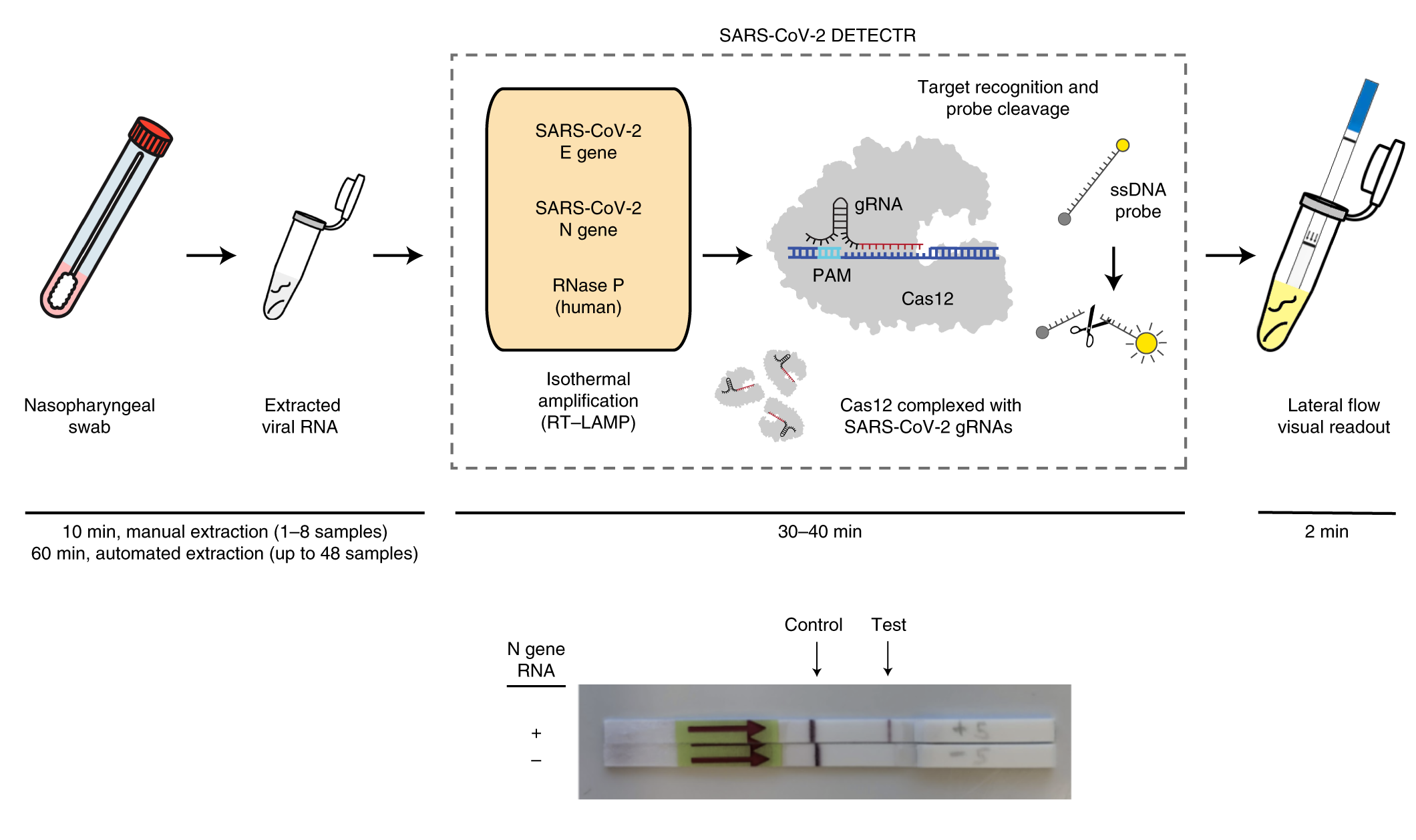
A lateral-flow módszer ennek az ssDNS-nek a detektálására használható. A reakcióelegybe belógatott szűrőpapír-jellegű szalag a kapilláris hatás segítségével elkezdi magába szívni a folyadékot és vele az ssDNS-riportert is. Az ssDNS egyik oldalára egy nagyméretű biotin molekulát kapcsolnak, a másik végére pedig egy FITC nevű molekulát – a lényeg, hogy mindegyiket nagyon specifikusan meg tudjuk köti. A biotint például sztreptavidinnal, ami a lateral-flow csíkok aljához közel található, ezért a biotin és vele kapcsolatban levő részek csak eddig fognak vándorolni. Ha nem történt hasítás a Cas12 által, akkor a FITC is, különben az tovább tud haladni a szalag mikrokapillárisaiban.
A detektálás során megjelenő csíkokat a belógatás után 2-3 perccel a FITC-et felismerő, arany nanorészecskék hozzák majd létre a hozzájuk kapcsolt ellenanyagok segítségével. Ha nem volt Cas12-hasítás (vagyis nem volt jelen a vírus), a szalag aljához közel, ha pedig jelen volt a vírus, attól távolabb jelennek meg elszíneződések.
Ha az amerikai gyógyszerhatóság által jóváhagyott SHERLOCK-rendszer beváltja a hozzá fűzött reményeket, akkor számíthatunk rá, hogy a közeljövőben hasonló rendszerek igen gyorsan elterjedhetnek – bár valószínűsíthető, hogy egyelőre inkább csak a klasszikusabb, PCR-alapú vírusdiagnosztika kiegészítőjeként, mintsem helyettesítőjeként.
(A poszt Kaszás Diána esszéje alapján készült. A borítókép a Broad Institute oldaláról származik, a lateral-flow működési mechanizmusát bemutató pedig a MileniaBiotec oldalról.)
Broughton JP, Deng X, Yu G, Fasching CF, Singh J et al., 2020, CRISPR–Cas12-based detection of SARS-CoV-2. Nat Biotechnol https://doi.org/10.1038/s41587-020-0513-4
Curti L, Pereyra-Bonnet F, Gimenez CA, 2020, An ultrasensitive, rapid, and portable coronavirus SARS-CoV-2 sequence detection method based on CRISPR-Cas12, bioRxiv
Gootenberg JS, Abudayyeh OO, Lee JW, Essletzbichler P, Dy AJ, et al., 2017, Nucleic acid detection with CRISPR-Cas13a/C2c2. Science 356, 438–442
Gootenberg JS, Abudayyeh OO, Kellner MJ, Joung J, Collins JJ and Zheng F, 2018, Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and Csm6 Science 360, 439-444
Wang H,Russa ML, Qi LS, 2016, CRISPR/Cas9 in Genome Editing and Beyond, Annual Review of Biochemistry, 85:227-264
Chen JS, Ma E, Harrington LB, Da Costa M, Tian X, et al., 2018, CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity, Science 360, 436-439
Garneau JE, Dupuis M-È, Villion M, Romero DA, Barrangou R, et al., 2010, The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA, Nature, 468, 67–71
Kocak DD, Gersbach CA 2018,From CRISPR scissors to virus sensors Nature 557, 168-169
Kostyusheva, A.; Brezgin, S.; Babin, Y.; Vasil’eva, I.; Kostyushev, D.; Chulanov, V., 2020, CRISPR-Cas Systems for Diagnosing Infectious Diseases. Preprints
M.A. Marra, S.J. Jones, C.R. Astell, R.A. Holt, A. Brooks-Wilson, Y.S. Butterfield, et al., 2003, The Genome sequence of the SARS-associated coronavirus, Science, 300 (2003), pp. 1399-1404
Notomi, T., Okayama, H., Masubuchi, H., et al., 2000, Loop-mediated isothermal amplification of DNA. Nucleic Acids Res., 2000, 28(12), e63
Wan Beom Park, Nak-Jung Kwon, Su-Jin Choi, Chang Kyung Kang, Pyoeng Gyun Choe, Jin Yong Kim, Jiyoung Yun, Gir-Won Lee, Moon-Woo Seong, Nam Joong Kim, Jeong-Sun Seo, and Myoung-don Oh, 2020 Virus Isolation from the First Patient with SARS-CoV-2 in Korea, The Korean Academy of Medical Sciences.
World Health Organization (WHO), 2003, Summary table of SARS cases by country, 1 November 2002–7 August 2003, Wkly. Epidemiol. Rec., 78 (2003), pp. 310-311

Nagyon erdekes, de ez most meg szakmabelikent is nehez olvasmany volt, a reszleteket most sem ertem.
Arrol lenne szo, hogy ha CRISPR megtalalja a targetet, akkor nem csak azt vagja, hanem altalanos nukleazkent mindent hasogat, ami a kozelebe kerul?
@fordulo_bogyo: Igen, ez egy különleges tulajdonsága a Cas12 és Cas13 fehérjéknek. (Pontosabban nem mindent, hanem csak ssDNS-t, vagy ssRNS-t, de azt szekvencia-specificitás nélkül).
@dolphin: Koszonom!