Nyilván a biológia egyik alapvető kérdése, hogyan alakulnak ki új gének az evolúció során? Az egyik legkorábban felismert modell egyszerűen azt tételezi fel, hogy egy működő gén duplikálódik, majd az egyikben mutációk történnek, amelyek egy új működés ellátására teszik alkalmassá. Ennek a modellnek az egyik gyenge pontja, hogy a tandem duplikációk általában evolúciós mértékkel mérve rövid ideig maradnak fenn, hiszen így egy gén két példányának kellene megmaradnia, holott szelekciós nyomás csak egy példányra hat, a második példány elvesztése nem okoz hátrányt az élőlénynek. Joakim Näsvall és munkatársai ennek a régi modellnek egy egészen picit módosított változatát közölték le nemrég, amelyben úgy képződhet egy új gén, hogy végig folyamatosan pozitív szelekciós nyomás hat a duplikált gén mindkét példányára.
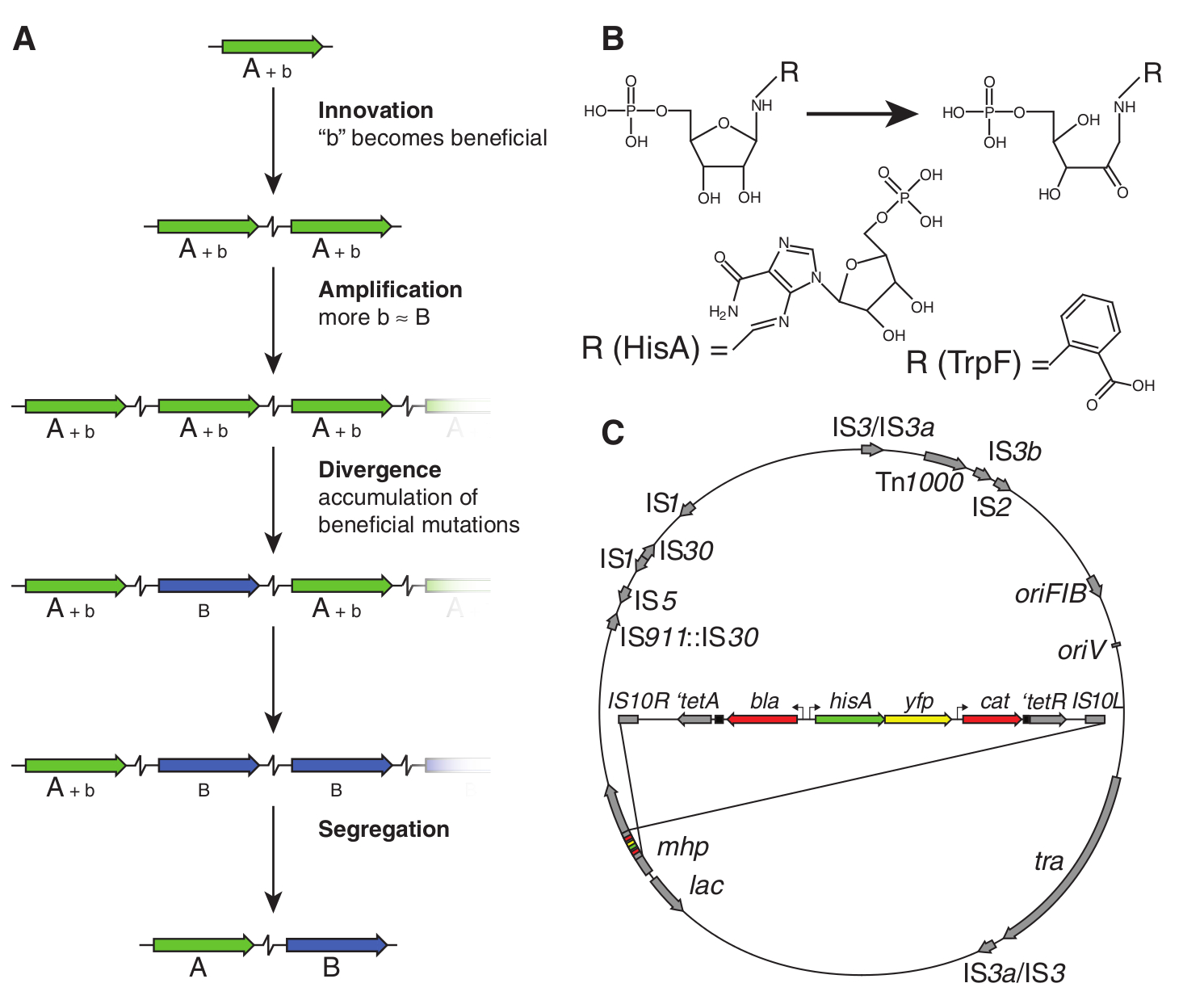
A modellt IAD -nek (innovation-amplification-divergence) nevezték el, annyiban különbözik a régebbi modelltől, hogy az ősi gén által kódolt fehérjének nem csak egy biokémai aktivitását tételezi fel, hanem egy másodlagos, kisebb hatékonyságú működést is. Ha a másodlagos működés is hasznos az élőlény számára, akkor ezt a működést javítja egy duplikáció, hiszen kétszer annyi géntermék nyilván kétszer annyi enzimaktivitást is jelent a sejtben. Így mindkét kópia fennmaradására szelekciós nyomás hat, hiszen ha bármelyik működésképtelenné válik, feleződik a géntermék mennyisége, így hosszú időn át fennmaradhat egy génnek több példánya is. Mivel azonban mindkét példány egymástól függetlenül evolvál, bármelyikben történhetnek előnyös mutációk, így könnyen előfordulhat, hogy az egyik példányban az egyik működést javító mutációk történnek, a másikban a másikat javító változások szaporodnak fel. Ez látható az első “a” ábrán.
De hogy ne csak elméleteket gyártsanak, egy kísérletet is terveztek a feltételezéseik igazolására Salmonella enterica baktériumokkal. A hisztidin aminosav (hisA) és a triptofán aminosav (trpF) bioszintéziséhez szükséges két enzimet kódoló gént vizsgálták. Mindkettő foszforibozil szubsztrát izomerizálódását katalizálja, de különböző szubsztráton működnek, így különböző terméket állítanak elő (első “b” ábra). Ha a baktériumsejteket minimáltáptalajon tartják, a sejtnek mindkét enzimre szüksége lesz, hogy előállíthassa a szükséges aminosavakat. Egy trpF mutáns törzset minimál táptalajra szélesztve azonosítottak egy olyan mutánst, aminek a HisA enzimje kis hatékonysággal képes ellátni a TrpF enzim feladatát is. Ebben a génben a 13-15 kodonok duplikációja és a 10. kodon pontmutációja kellett ehhez, ami az enzimben a 10. aminosav Asp -> Gly cseréjét eredményezi. Ezt a gént egy konstitutív promóterrel egészítették ki és a sárgán fluoreszkáló fehérjét (YFP) kódoló génnel (első “c” ábra), az egészet egy kivételesen alacsony kópiaszámú plazmidba klónozták, amely átlagosan két példányban van jelen a sejtben, majd a plazmidot egy hisA és trpF mutánsba juttatták, így a sejt a bejuttatott gén nélkül egyik aminosavat sem volt képes előállítani. Ez a baktériumtörzs minimáltáptalajon ~5,1 óra alatt tudott osztódni, triptofánnal kiegészített táptalajon ~2,8 óra alatt, hisztidinnel kiegészített táptalajon ~2,6 óra alatt, mindkét aminosav jelenlétében pedig ~1,5 óra alatt.
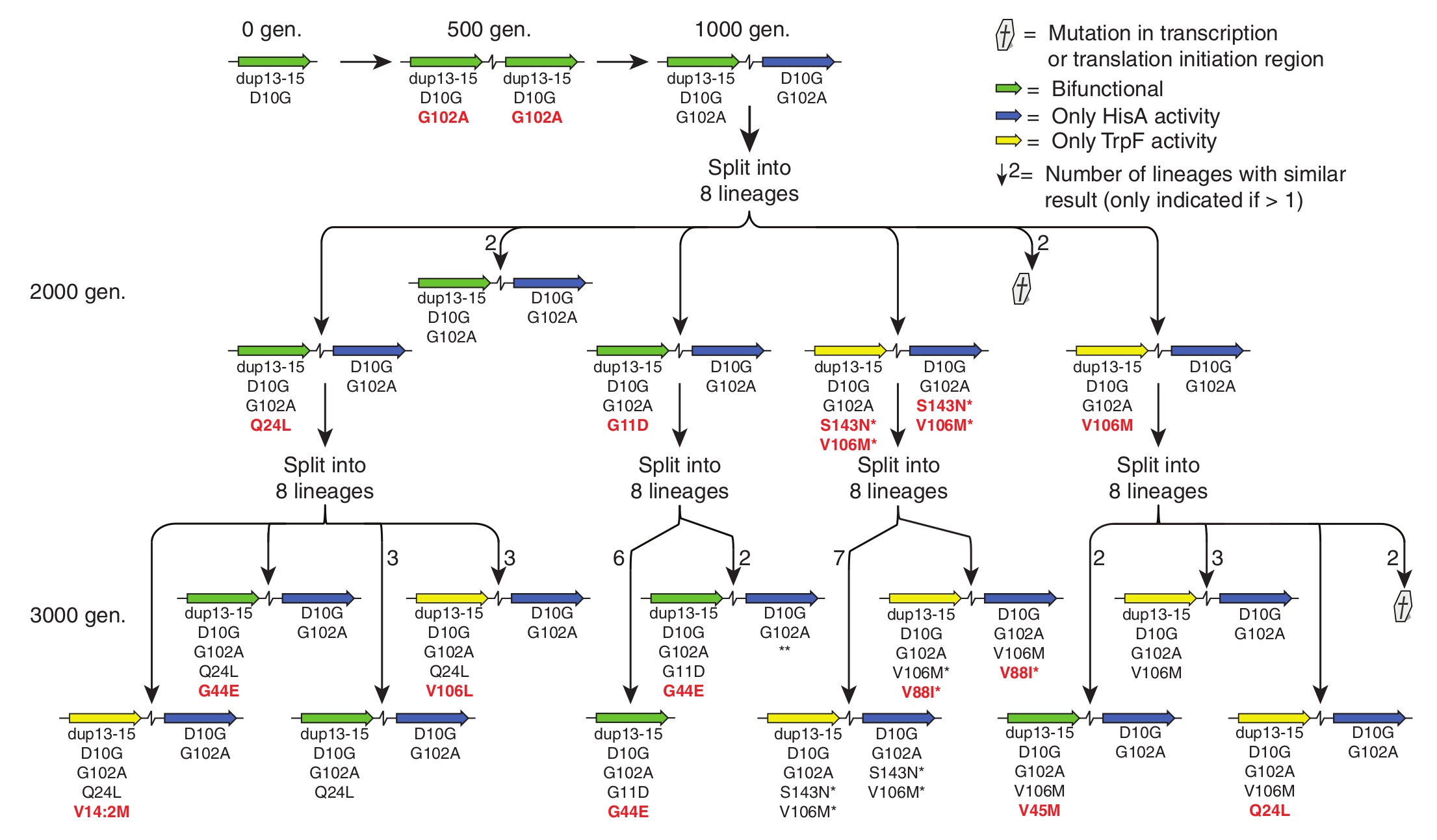
Ebből a baktériumtörzsből több különböző, egymástól független kultúrát indítottak, aztán ezekkel semmit sem csináltak, csak hagyták őket minimál táptalajon evolválni, ennek eredménye látható a második ábrán. (A cikk szövegében azt írják, hogy az eredeti törzsből indították a különböző kultúrákat, de az ábrán úgy tűnik, hogy ezer nemzedéknyi evolúció után oltották le őket, ez eléggé zavaró. ) Zöld nyilak jelzik a mindkét aktivitással rendelkező, kék nyilak a HisA, sárga nyilak a TrpF aktivitású fehérjéket kódoló géneket. Néhány száz nemzedék alatt az osztódáshoz szükséges idő 5,1 óráról törzstől függően 1,9 – 2,5 órára csökkent, nem meglepő módon ezzel együtt a hisA gén kifejeződése is lépésenként emelkedett, akár hússzorosára, a hisA gént tartalmazó szakasz duplikációival. Háromezer nemzedéknyi evolúció során minden egyes törzs a növekedését gyorsító mutációkat szedett össze, számos törzsben megtörtént a modell által előre jelzett eredmény is, a duplikálódott gének egyike az egyik enzimaktivitásra, másika a másik enzimaktivitásra specializálódott.
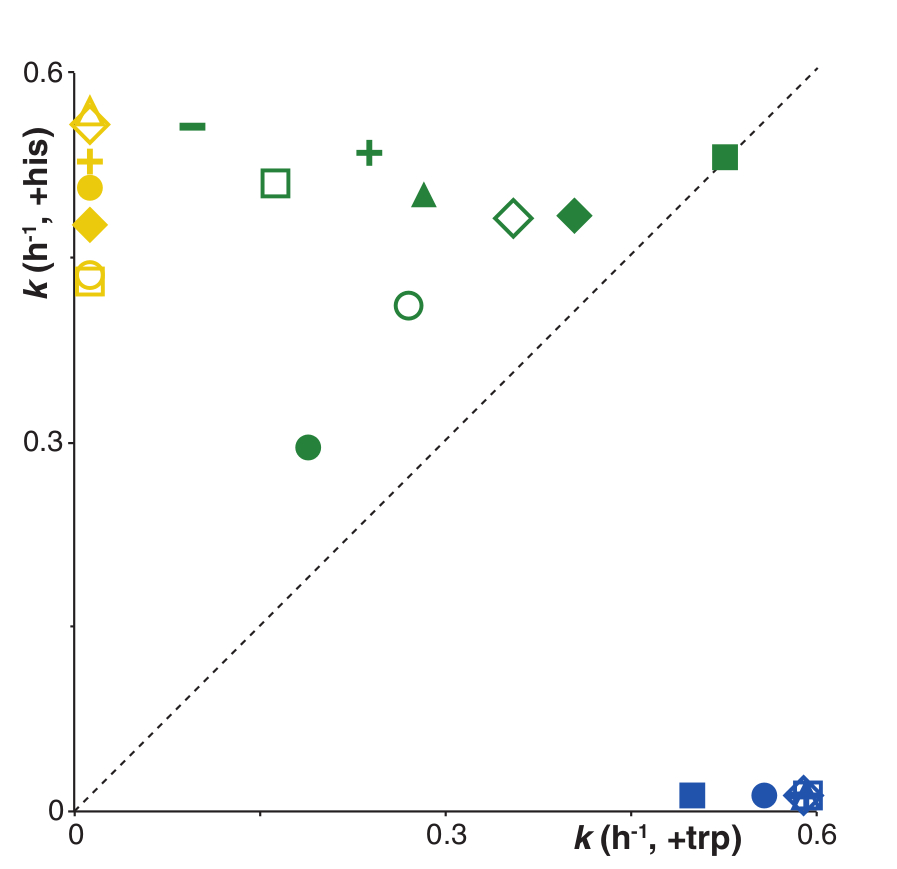
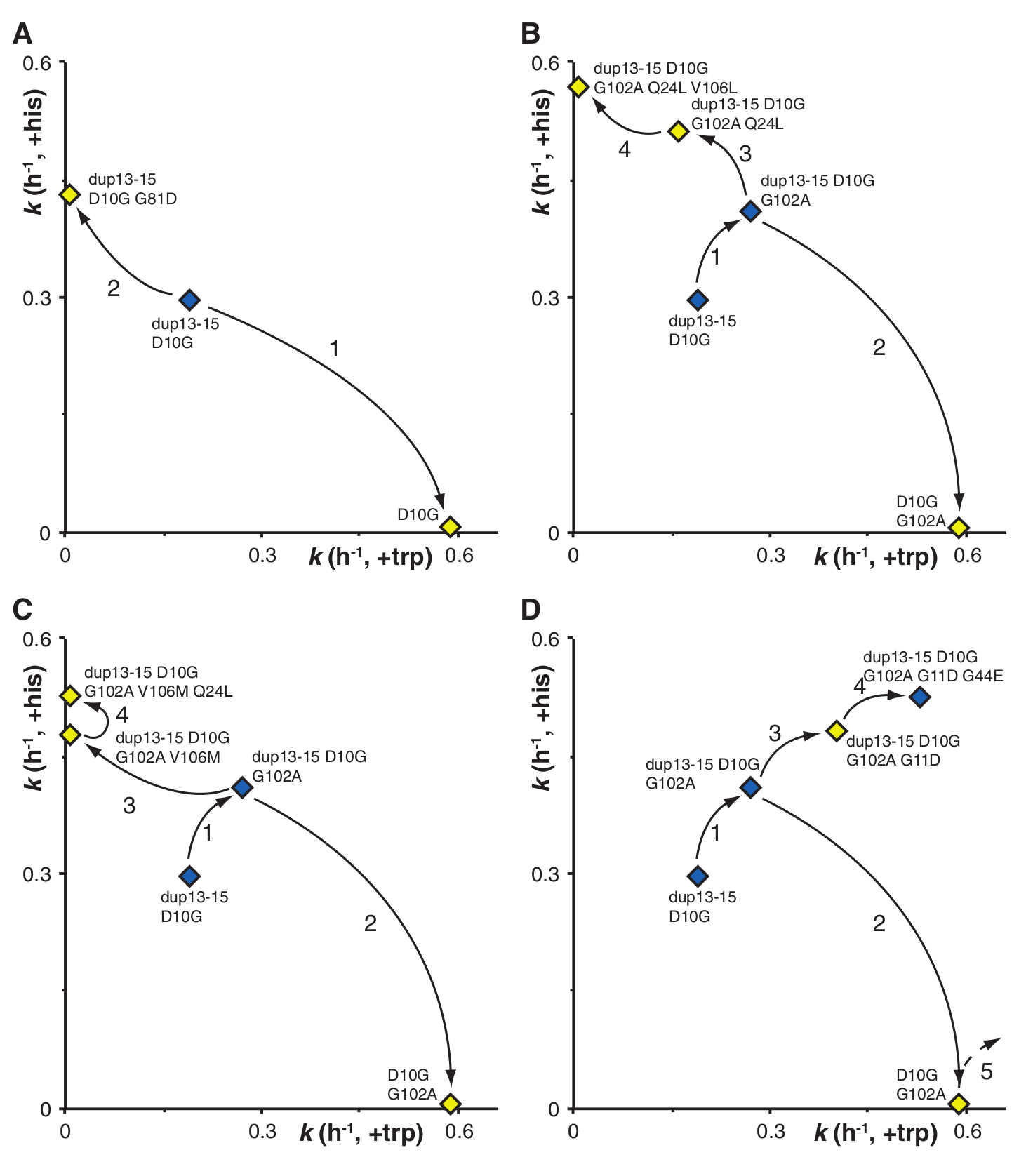
Ez után fogtak huszonkét függetlenül evolvált gént és egyesével egy teljesen más baktériumtörzs genomjába juttatták őket, hogy kizárhassák a genom más pontjain történt háttérmutációk hatását és mindegyik gén egyetlen példányban legyen a sejtben, majd ezen baktériumtörzsek viselkedését vizsgálták meg minimáltáptalajon. Az eredmény a harmadik ábrán látható, a függőleges tengelyen a hisztidinnel, a vízszintes tengelyen a triptofánnal kiegészített minimáltáptalajon mért osztódás sebességet ábrázolták, mint látható, a legtöbb enzim csak az egyik működést végzi jól, ám azért találtak egy-két olyan enzimet is, amelyik továbbra is mindkét aktivitással rendelkezik, ezeket jelölték zölddel. Több esetben egyetlen sejtből származott a két különböző működésű enzim, elég jól mutatva a modell életképességét, ebben az esetben ugyanis egy ősi enzimet kódoló gén duplikációja nyomán a leánygénekben olyan mutációk történtek, amelyek nyomán két különböző aktivitású enzimet kódolnak. Ebből megállapítható, hogy az IAD modell a valóságban is működik, megfelelő szelekciós nyomás hatására egy enzimet kódoló génből viszonylag gyorsan keletkezik két, egymástól eltérő gén. Az utolsó ábrán négy baktériumtörzs történetét mutatják be a szerzők, az “a” estben egy generalista enzimből két specialista alakult ki, a “b” és “c” esetben előbb a kétféle aktivitású enzim működése javult, majd ebből alakult ki két egyetlen aktivitással rendelkező enzim, a “d” ábrán pedig egy mindkét aktivitással rendelkező enzim kialakulása követhető, egyes mutációkra lebontott részletességgel.
Näsvall J, Sun L, Roth JR, Andersson DI (2012) Real-time evolution of new genes by innovation, amplification and divergence. Science. 338. 384-387

A nyáron volt egy Nature cikk is, ahol az élesztőben néztek valami hasonlót, igaz, ott a funkciót nem nagyon feszegették.
http://www.nature.com/nature/journal/vaop/ncurrent/full/nature11184.html